Диаграммы Венна с R? [закрытый]
другие пакеты для выполнения диаграмм Venn в R помимо пакета limma.
У кого-нибудь есть советы?
вот заметки о выполнении диаграмм Венна с пакетами limma.
8 ответов
у Дункана Мердока есть пакет venn, который не находится на CRAN. (наконечник шляпы Габору Гротендику)
вы также можете прочитать об этом в "журнале статистического программного обеспечения".
здесь Vennerable пакет на R-forge.
source("http://bioconductor.org/biocLite.R")
biocLite(c("graph", "RBGL", "gtools", "xtable"))
install.packages("Vennerable", repos="http://R-Forge.R-project.org")
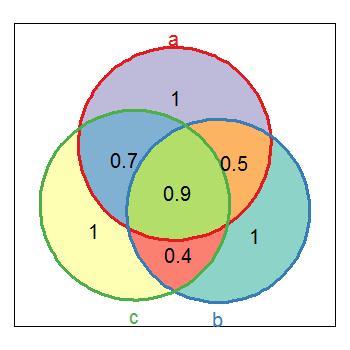
Я использую две пользовательские функции, которые на хитрость. Моя реализация venndia строит диаграмму venn и возвращает списки перекрытий между A и B (и C). См. код ниже.
С этими, вы можете
vd <- venndia(A=LETTERS[1:15], B=LETTERS[5:20], getdata=TRUE)
который будет как строить, так и возвращать данные. вы можете отключить возврат данных
venndia(A=LETTERS[1:15], B=LETTERS[5:20])
поскольку getdata по умолчанию FALSE. / Даниэль
circle <- function(x, y, r, ...) {
ang <- seq(0, 2*pi, length = 100)
xx <- x + r * cos(ang)
yy <- y + r * sin(ang)
polygon(xx, yy, ...)
}
venndia <- function(A, B, C, getdata=FALSE, ...){
cMissing <- missing(C)
if(cMissing){ C <- c() }
unionAB <- union(A, B)
unionAC <- union(A, C)
unionBC <- union(B, C)
uniqueA <- setdiff(A, unionBC)
uniqueB <- setdiff(B, unionAC)
uniqueC <- setdiff(C, unionAB)
intersAB <- setdiff(intersect(A, B), C)
intersAC <- setdiff(intersect(A, C), B)
intersBC <- setdiff(intersect(B, C), A)
intersABC <- intersect(intersect(A, B), intersect(B, C))
nA <- length(uniqueA)
nB <- length(uniqueB)
nC <- length(uniqueC)
nAB <- length(intersAB)
nAC <- length(intersAC)
nBC <- length(intersBC)
nABC <- length(intersABC)
par(mar=c(2, 2, 0, 0))
plot(-10, -10, ylim=c(0, 9), xlim=c(0, 9), axes=FALSE, ...)
circle(x=3, y=6, r=3, col=rgb(1,0,0,.5), border=NA)
circle(x=6, y=6, r=3, col=rgb(0,.5,.1,.5), border=NA)
circle(x=4.5, y=3, r=3, col=rgb(0,0,1,.5), border=NA)
text( x=c(1.2, 7.7, 4.5), y=c(7.8, 7.8, 0.8), c("A", "B", "C"), cex=3, col="gray90" )
text(
x=c(2, 7, 4.5, 4.5, 3, 6, 4.5),
y=c(7, 7, 2 , 7 , 4, 4, 5),
c(nA, nB, nC, nAB, nAC, nBC, nABC),
cex=2
)
if(getdata){
list(A=uniqueA, B=uniqueB, C=uniqueC,
AB=intersAB , AC=intersAC , BC=intersBC ,
ABC=intersABC
)
}
}
это происходит очень поздно, но это может быть полезно для других, ищущих ответ: VennDiagram, на кран здесь.
Он позволяет несколько наборов (четыре набора для venn, 3 набора для диаграмм Эйлера), настраиваемые цвета и шрифты, простой синтаксис и и лучше всего размер кругов пропорционален размеру наборов данных (по крайней мере, при сравнении 2 наборов данных). Чтобы установить:
install.packages("VennDiagram")
library(VennDiagram)
для тех, кто использует курсе bioconductor пакеты и работая с геномными координатами, недавно венндиаграмма была реализовала в пакете ChIPpeakAnno (версия 2.5.12) и позволяет довольно пересечениям геномных координат, например, пиков Chip-seq. Для ранних усыновителей вам может потребоваться установить пакета разработки.
peaks1 = RangedData(IRanges(start = c(967654, 2010897, 2496704),
end = c(967754, 2010997, 2496804), names = c("Site1", "Site2", "Site3")),
space = c("1", "2", "3"), strand=as.integer(1),feature=c("a","b","f"))
peaks2 = RangedData(IRanges(start = c(967659, 2010898,2496700,3075866,3123260),
end = c(967869, 2011108, 2496920, 3076166, 3123470),
names = c("t1", "t2", "t3", "t4", "t5")),
space = c("1", "2", "3", "1", "2"), strand = c(1, 1, -1,-1,1), feature=c("a","b","c","d","a"))
makeVennDiagram(RangedDataList(peaks1,peaks2, peaks1, peaks2), NameOfPeaks=c("TF1", "TF2","TF3", "TF4"),
totalTest=100,useFeature=TRUE, main="Venn Diagram",
col = "transparent",fill = c("cornflowerblue", "green", "yellow", "darkorchid1"),
alpha = 0.50,label.col = c("orange", "white", "darkorchid4", "white", "white", "white", "white", "white", "darkblue", "white", "white", "white", "white", "darkgreen", "white"), cat.col = c("darkblue", "darkgreen", "orange", "darkorchid4"))
вот ссылка на другую версию для 3-переменные данные: http://elliotnoma.wordpress.com/2011/02/09/venn-diagram/
код также доступен в пакете colorfulVennPlot: http://cran.r-project.org/web/packages/colorfulVennPlot/index.html
Я бы рекомендовал пакет VennDiagram: http://cran.r-project.org/web/packages/VennDiagram/VennDiagram.pdf
на pake 19 Вы найдете 10 pake очень хороших примеров (как продвинутых, так и упрощенных). До сих пор я не нашел ничего, что он не может сделать, что мне нужно.
