ggplot2 boxplots-Как избежать дополнительного вертикального пространства, когда нет значительных сравнений?
после многих вопросов о том, как сделать boxplots с гранями и уровнями значимости, особенно этой и этой, у меня еще одна маленькая проблема.
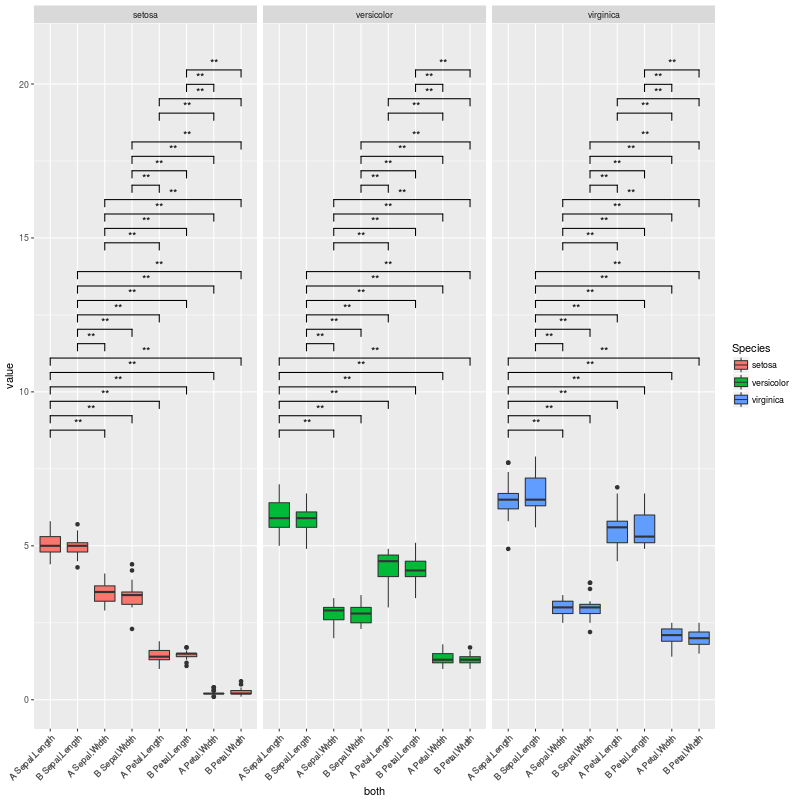
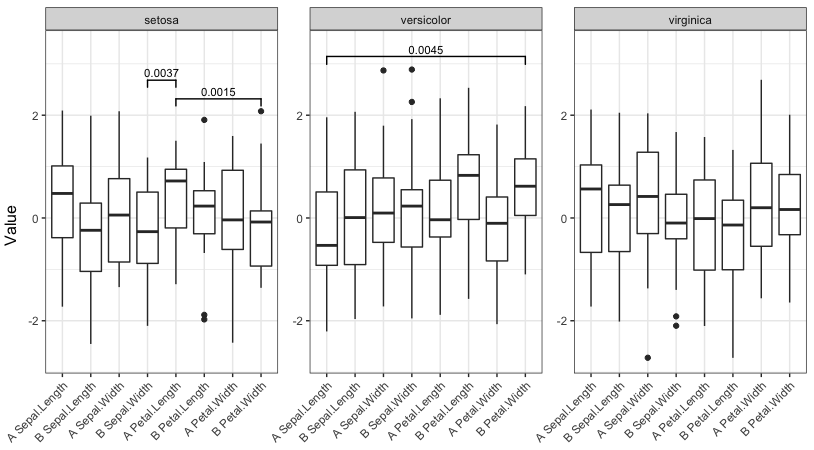
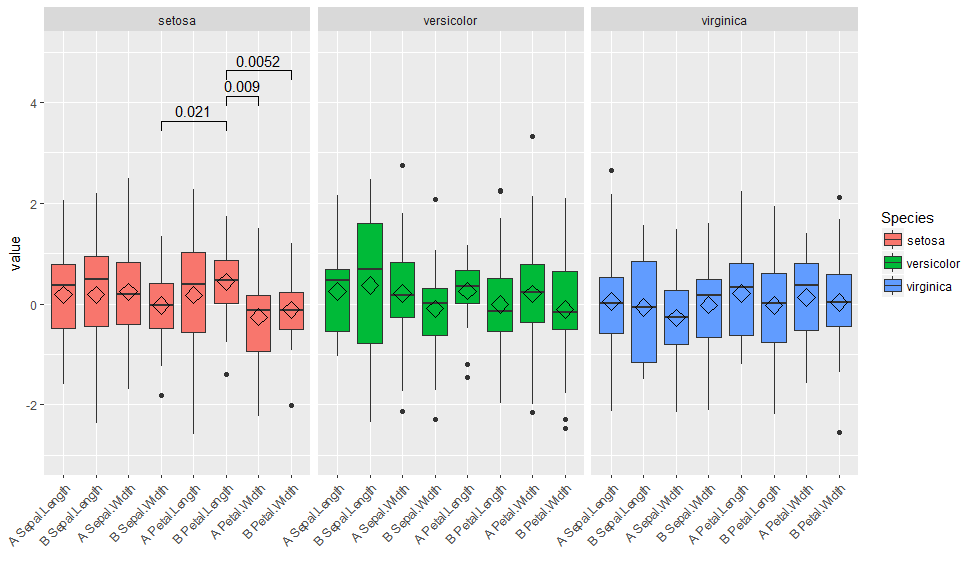
мне удалось создать сюжет, показанный ниже, что именно то, что я хочу.
проблема, с которой я сталкиваюсь сейчас, - это когда у меня очень мало или нет значительных сравнений; в этих случаях все пространство, посвященное скобкам, показывающим уровни значимости, все еще сохраняется, но я хочу от него избавиться.
пожалуйста, проверьте это MWE с набором данных iris:
library(reshape2)
library(ggplot2)
data(iris)
iris$treatment <- rep(c("A","B"), length(iris$Species)/2)
mydf <- melt(iris, measure.vars=names(iris)[1:4])
mydf$treatment <- as.factor(mydf$treatment)
mydf$variable <- factor(mydf$variable, levels=sort(levels(mydf$variable)))
mydf$both <- factor(paste(mydf$treatment, mydf$variable), levels=(unique(paste(mydf$treatment, mydf$variable))))
a <- combn(levels(mydf$both), 2, simplify = FALSE)#this 6 times, for each lipid class
b <- levels(mydf$Species)
CNb <- relist(
paste(unlist(a), rep(b, each=sum(lengths(a)))),
rep.int(a, length(b))
)
CNb
CNb2 <- data.frame(matrix(unlist(CNb), ncol=2, byrow=T))
CNb2
#new p.values
pv.df <- data.frame()
for (gr in unique(mydf$Species)){
for (i in 1:length(a)){
tis <- a[[i]] #variable pair to test
as <- subset(mydf, Species==gr & both %in% tis)
pv <- wilcox.test(value ~ both, data=as)$p.value
ddd <- data.table(as)
asm <- as.data.frame(ddd[, list(value=mean(value)), by=list(both=both)])
asm2 <- dcast(asm, .~both, value.var="value")[,-1]
pf <- data.frame(group1=paste(tis[1], gr), group2=paste(tis[2], gr), mean.group1=asm2[,1], mean.group2=asm2[,2], log.FC.1over2=log2(asm2[,1]/asm2[,2]), p.value=pv)
pv.df <- rbind(pv.df, pf)
}
}
pv.df$p.adjust <- p.adjust(pv.df$p.value, method="BH")
colnames(CNb2) <- colnames(pv.df)[1:2]
# merge with the CN list
pv.final <- merge(CNb2, pv.df, by.x = c("group1", "group2"), by.y = c("group1", "group2"))
# fix ordering
pv.final <- pv.final[match(paste(CNb2$group1, CNb2$group2), paste(pv.final$group1, pv.final$group2)),]
# set signif level
pv.final$map.signif <- ifelse(pv.final$p.adjust > 0.05, "", ifelse(pv.final$p.adjust > 0.01,"*", "**"))
# subset
G <- pv.final$p.adjust <= 0.05
CNb[G]
P <- ggplot(mydf,aes(x=both, y=value)) +
geom_boxplot(aes(fill=Species)) +
facet_grid(~Species, scales="free", space="free_x") +
theme(axis.text.x = element_text(angle=45, hjust=1)) +
geom_signif(test="wilcox.test", comparisons = combn(levels(mydf$both),2, simplify = F),
map_signif_level = F,
vjust=0.5,
textsize=4,
size=0.5,
step_increase = 0.06)
P2 <- ggplot_build(P)
#pv.final$map.signif <- "" #UNCOMMENT THIS LINE TO MOCK A CASE WHERE THERE ARE NO SIGNIFICANT COMPARISONS
#pv.final$map.signif[c(1:42,44:80,82:84)] <- "" #UNCOMMENT THIS LINE TO MOCK A CASE WHERE THERE ARE JUST A COUPLE OF SIGNIFICANT COMPARISONS
P2$data[[2]]$annotation <- rep(pv.final$map.signif, each=3)
# remove non significants
P2$data[[2]] <- P2$data[[2]][P2$data[[2]]$annotation != "",]
# and the final plot
png(filename="test.png", height=800, width=800)
plot(ggplot_gtable(P2))
dev.off()
который производит этот сюжет:
сюжет выше именно то, что я хочу... Но я сталкиваюсь со случаями, когда нет значительных сравнений или очень мало. В этих случаях много вертикального пространства остается пустым.
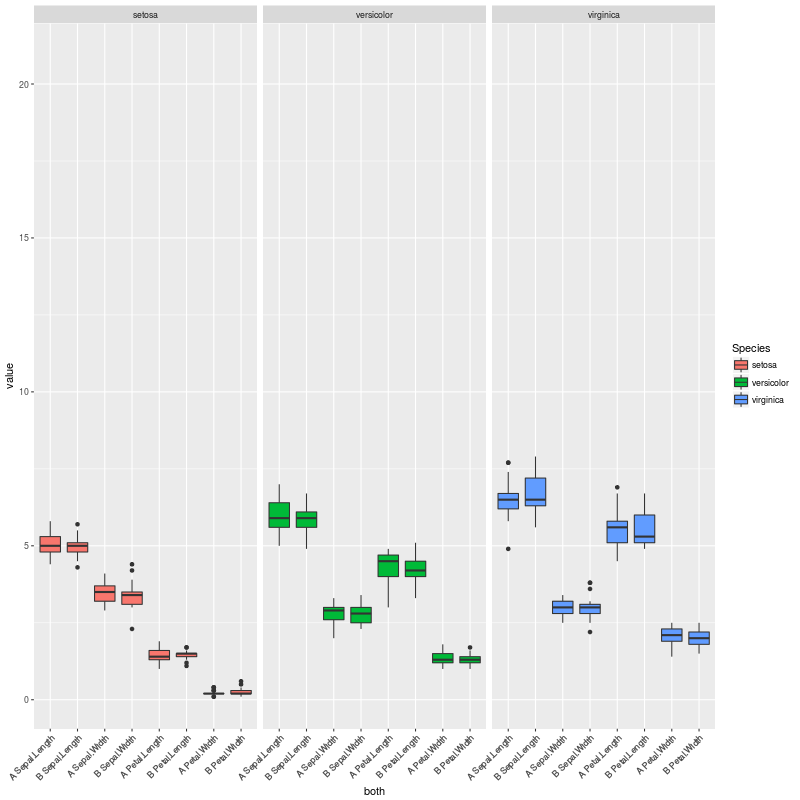
чтобы проиллюстрировать эти сценарии, мы можем раскомментировать строку:
pv.final$map.signif <- "" #UNCOMMENT THIS LINE TO MOCK A CASE WHERE THERE ARE NO SIGNIFICANT COMPARISONS
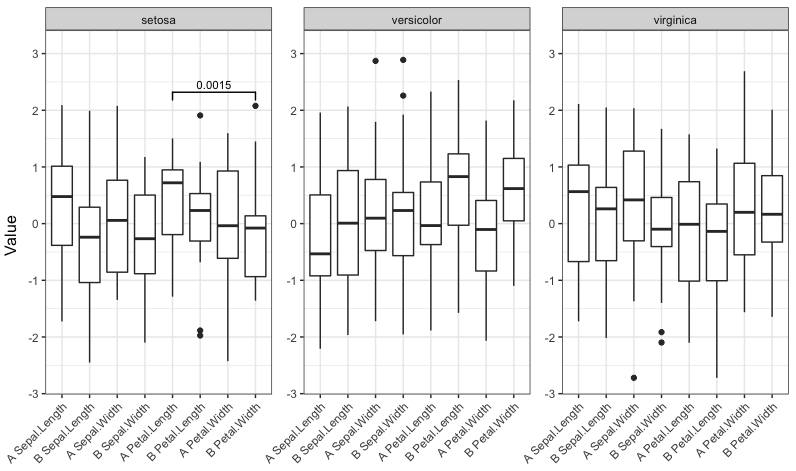
так когда нет никаких существенных сравнений, я получаю этот сюжет:
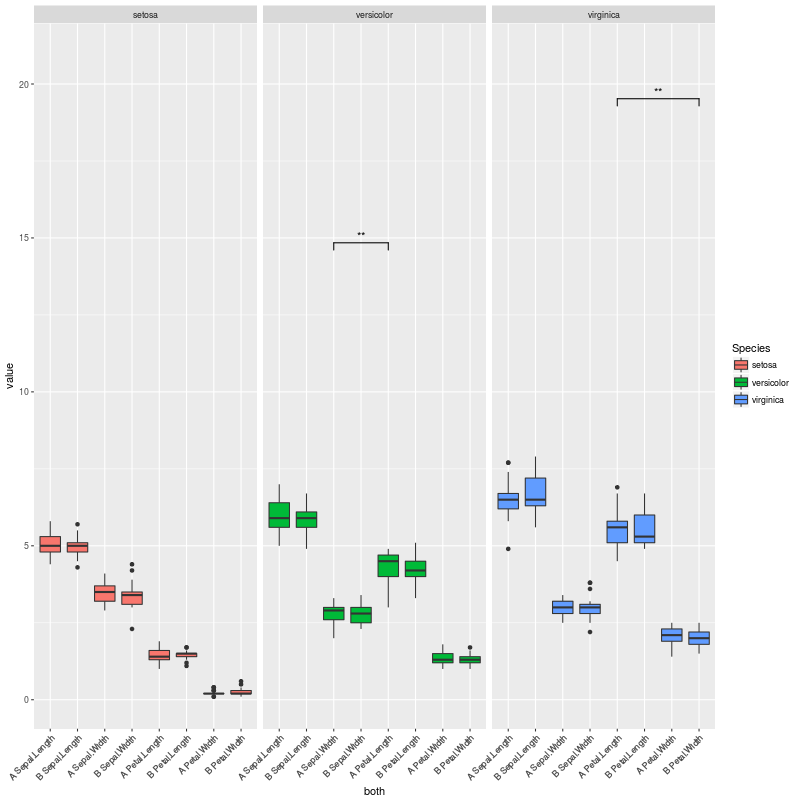
если мы раскомментируем эту другую строку:
pv.final$map.signif[c(1:42,44:80,82:84)] <- "" #UNCOMMENT THIS LINE TO MOCK A CASE WHERE THERE ARE JUST A COUPLE OF SIGNIFICANT COMPARISONS
мы находимся в случае, когда есть только пара значимых сравнений, и получаем этот сюжет:
Итак, мой вопрос здесь:
как настроить вертикальное пространство на количество значимых сравнений, поэтому вертикального пространства не осталось там?
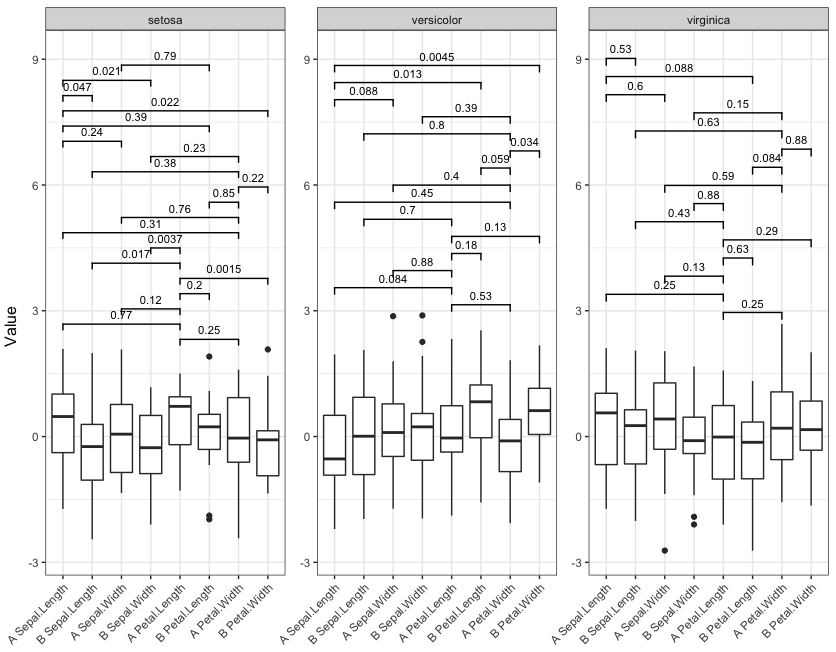
там может быть что-то, что я мог бы изменить в step_increase или y_position внутри geom_signif(), поэтому я оставляю место только для значительных сравнений в CNb[G]...
2 ответов
один из вариантов-предварительно вычислить p-значения для каждой комбинации both уровни, а затем выберите только значимые для построения графика. Поскольку мы знаем, сколько из них значимо, мы можем настроить y-диапазоны графиков для учета этого. Однако это не похоже на geom_signif способен выполнять только вычисления в пределах фасета для аннотаций p-значения (см. справку для выложить отдельные участки, как если бы они были гранеными.
(чтобы ответить на комментарии, я хочу подчеркнуть, что все сюжеты по-прежнему создаются с помощью ggplot2, но мы создаем то, что было бы тремя фасетными панелями одного сюжета как три отдельных участка а затем разложите их вместе, как будто они были огранены.)
функции ниже жестко для фрейма данных и имен столбцов в OP, но, конечно, можно обобщить, чтобы взять любые имена фреймов данных и столбцов.
library(gridExtra)
library(tidyverse)
# Change data to reduce number of statistically significant differences
set.seed(2)
df = mydf %>% mutate(value=rnorm(nrow(mydf)))
# Function to generate and lay out the plots
signif_plot = function(signif.cutoff=0.05, height.factor=0.23) {
# Get full range of y-values
y_rng = range(df$value)
# Generate a list of three plots, one for each Species (these are the facets)
plot_list = lapply(split(df, df$Species), function(d) {
# Get pairs of x-values for current facet
pairs = combn(sort(as.character(unique(d$both))), 2, simplify=FALSE)
# Run wilcox test on every pair
w.tst = pairs %>%
map_df(function(lv) {
p.value = wilcox.test(d$value[d$both==lv[1]], d$value[d$both==lv[2]])$p.value
data.frame(levs=paste(lv, collapse=" "), p.value)
})
# Record number of significant p.values. We'll use this later to adjust the top of the
# y-range of the plots
num_signif = sum(w.tst$p.value <= signif.cutoff)
# Plot significance levels only for combinations with p <= signif.cutoff
p = ggplot(d, aes(x=both, y=value)) +
geom_boxplot() +
facet_grid(~Species, scales="free", space="free_x") +
geom_signif(test="wilcox.test", comparisons = pairs[which(w.tst$p.value <= signif.cutoff)],
map_signif_level = F,
vjust=0,
textsize=3,
size=0.5,
step_increase = 0.08) +
theme_bw() +
theme(axis.title=element_blank(),
axis.text.x = element_text(angle=45, hjust=1))
# Return the plot and the number of significant p-values
return(list(num_signif, p))
})
# Get the highest number of significant p-values across all three "facets"
max_signif = max(sapply(plot_list, function(x) x[[1]]))
# Lay out the three plots as facets (one for each Species), but adjust so that y-range is same
# for each facet. Top of y-range is adjusted using max_signif.
grid.arrange(grobs=lapply(plot_list, function(x) x[[2]] +
scale_y_continuous(limits=c(y_rng[1], y_rng[2] + height.factor*max_signif))),
ncol=3, left="Value")
}
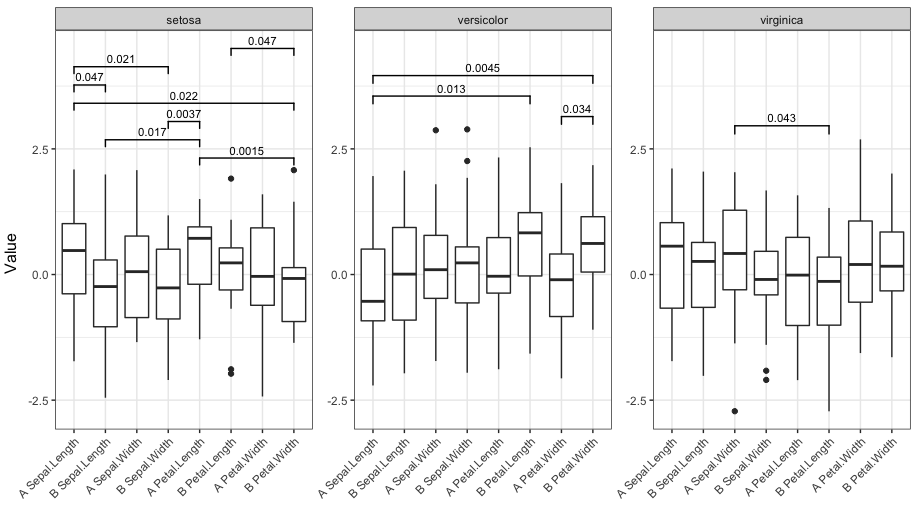
Теперь запустите функцию с четырьмя различными сокращениями значимости:
signif_plot(0.05)
signif_plot(0.01)
signif_plot(0.9)
signif_plot(0.0015)
вы можете попробовать. Хотя ответ похож на мой ответ здесь, я добавил Теперь функцию.
library(tidyverse)
library(ggsignif)
# 1. your data
set.seed(2)
df <- as.tbl(iris) %>%
mutate(treatment=rep(c("A","B"), length(iris$Species)/2)) %>%
gather(key, value, -Species, -treatment) %>%
mutate(value=rnorm(n())) %>%
mutate(key=factor(key, levels=unique(key))) %>%
mutate(both=interaction(treatment, key, sep = " "))
# 2. pairwise.wilcox.test for 1) validation and 2) to calculate the ylim
Wilcox <- df %>%
split(., .$Species) %>%
map(~tidy(pairwise.wilcox.test(.$value, .$both, p.adjust.method = "none"))) %>%
map(~filter(.,.$p.value < 0.05)) %>%
bind_rows(.id="Species") %>%
mutate(padjust=p.adjust(p.value, method = "BH"))
# 3. calculate y range
Ylim <- df %>%
summarise(Min=round(min(value)),
Max=round(max(value))) %>%
mutate(Max=Max+0.5*group_by(Wilcox, Species) %>% count() %>% with(.,max(n)))
%>% c()
# 4. the plot function
foo <- function(df, Ylim, Signif=0.05){
P <- df %>%
ggplot(aes(x=both, y=value)) +
geom_boxplot(aes(fill=Species)) +
facet_grid(~Species) +
ylim(Ylim$Min, Ylim$Max)+
theme(axis.text.x = element_text(angle=45, hjust=1)) +
geom_signif(comparisons = combn(levels(df$both),2,simplify = F),
map_signif_level = F, test = "wilcox.test" ) +
stat_summary(fun.y=mean, geom="point", shape=5, size=4) +
xlab("")
# 5. remove not significant values and add step increase
P_new <- ggplot_build(P)
P_new$data[[2]] <- P_new$data[[2]] %>%
filter(as.numeric(as.character(annotation)) < 0.05) %>%
group_by(PANEL) %>%
mutate(index=(as.numeric(group[drop=T])-1)*0.5) %>%
mutate(y=y+index,
yend=yend+index) %>%
select(-index) %>%
as.data.frame()
# the final plot
plot(ggplot_gtable(P_new))
}
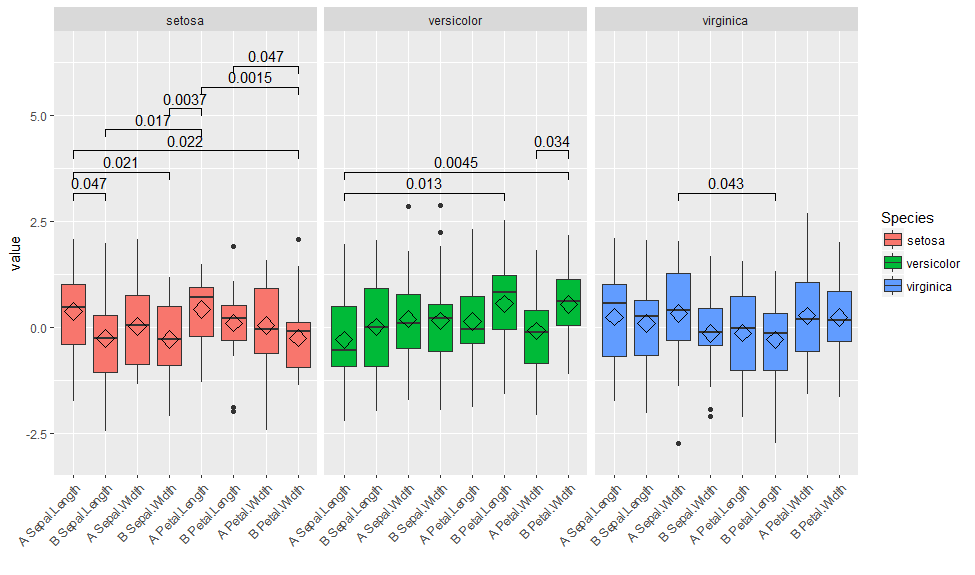
foo(df, Ylim)
попытка других данных
set.seed(12345)
df <- as.tbl(iris) %>%
mutate(treatment=rep(c("A","B"), length(iris$Species)/2)) %>%
gather(key, value, -Species, -treatment) %>%
mutate(value=rnorm(n())) %>%
mutate(key=factor(key, levels=unique(key))) %>%
mutate(both=interaction(treatment, key, sep = " "))
foo(df, list(Min=-3,Max=5))
конечно, вы можете добавить расчет Ylim в функцию. Кроме того, вы можете изменить или добавить ggtitel(), ylab() и изменить цвет.