Как дать sns.clustermap расстоянии матрицы вычислен?
обычно, когда я делаю дендрограммы и тепловые карты, я использую матрицу расстояний и делаю кучу SciPy вещи. Я хочу попробовать Seaborn но Seaborn хочет мои данные в прямоугольной форме (строки=образцы, cols=атрибуты, а не матрица расстояний)?
Я по существу хочу использовать seaborn в качестве бэкэнда для вычисления моей дендрограммы и прикрепления ее к моей тепловой карте. Возможно ли это? Если нет, Может ли это быть особенностью в будущем.
возможно, есть параметры, которые я могу отрегулируйте, чтобы он мог принимать матрицу расстояний вместо прямоугольной матрицы?
здесь использование:
seaborn.clustermap¶
seaborn.clustermap(data, pivot_kws=None, method='average', metric='euclidean',
z_score=None, standard_scale=None, figsize=None, cbar_kws=None, row_cluster=True,
col_cluster=True, row_linkage=None, col_linkage=None, row_colors=None,
col_colors=None, mask=None, **kwargs)
мой код ниже:
from sklearn.datasets import load_iris
iris = load_iris()
X, y = iris.data, iris.target
DF = pd.DataFrame(X, index = ["iris_%d" % (i) for i in range(X.shape[0])], columns = iris.feature_names)
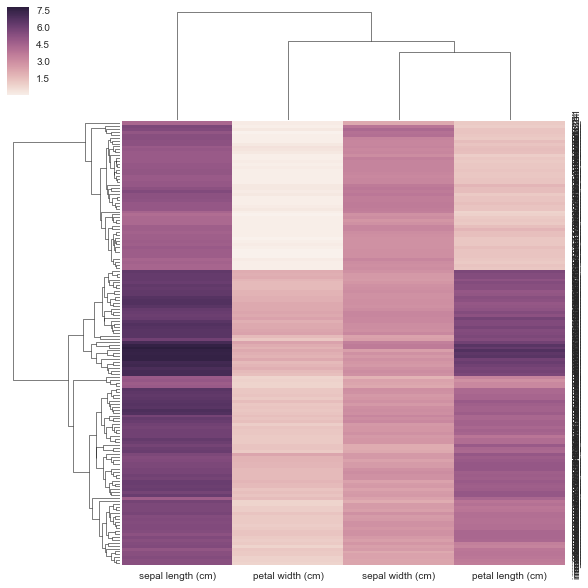
Я не думаю, что мой метод верен ниже, потому что я даю ему предварительно вычисленную матрицу расстояний, а не прямоугольную матрицу данных, как она запрашивает. Нет примеров того, как использовать матрицу корреляции/расстояния с clustermap но есть для https://stanford.edu / ~mwaskom/software/seaborn/examples/network_correlations.html но порядок не кластеризован с равниной sns.heatmap func.
DF_corr = DF.T.corr()
DF_dism = 1 - DF_corr
sns.clustermap(DF_dism)
1 ответов
вы можете передать предварительно вычисленную матрицу расстояний как связь с clustermap():
import pandas as pd, seaborn as sns
import scipy.spatial as sp, scipy.cluster.hierarchy as hc
from sklearn.datasets import load_iris
sns.set(font="monospace")
iris = load_iris()
X, y = iris.data, iris.target
DF = pd.DataFrame(X, index = ["iris_%d" % (i) for i in range(X.shape[0])], columns = iris.feature_names)
DF_corr = DF.T.corr()
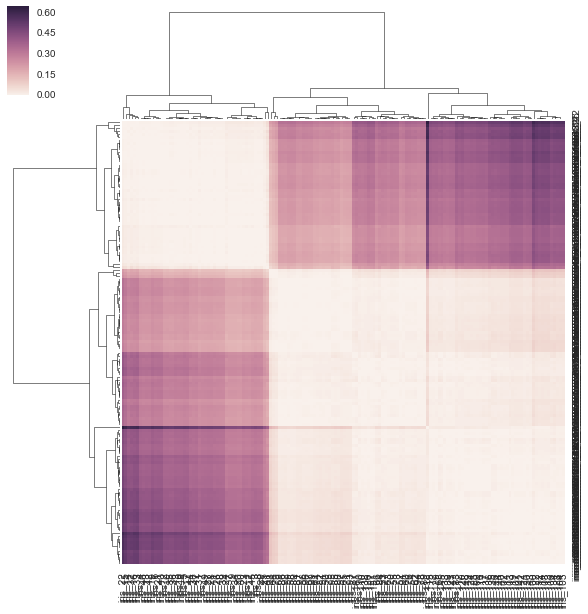
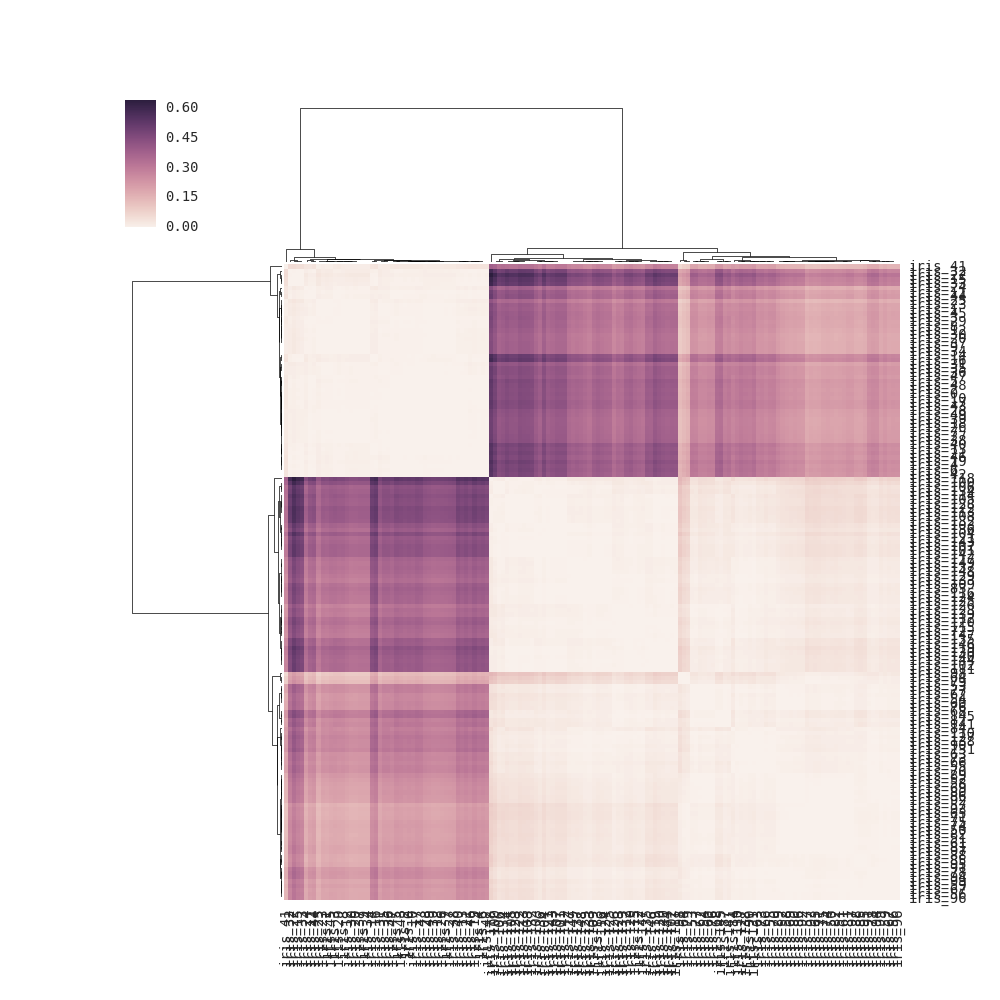
DF_dism = 1 - DF_corr # distance matrix
linkage = hc.linkage(sp.distance.squareform(DF_dism), method='average')
sns.clustermap(DF_dism, row_linkage=linkage, col_linkage=linkage)
на clustermap(distance_matrix) (т. е. без передачи связи) Связь вычисляется внутри на основе попарных расстояний строк и столбцов в матрице расстояний (см. Примечание ниже для получения полной информации) вместо использования элементов матрицы расстояний напрямую (правильное решение). В результате, выход Несколько отличается от того, в вопрос:
Примечание: если нет row_linkage перешло к clustermap(), связь строк определяется внутренне, рассматривая каждую строку как "точку" (наблюдение) и вычисляя попарные расстояния между точками. Таким образом, дендрограмма строк отражает сходство строк. Аналогично для col_linkage, где каждый столбец является точка. Это объяснение, вероятно, следует добавить к docs. Здесь первый пример docs изменен, чтобы сделать внутренний расчет связи явный:
import seaborn as sns; sns.set()
import scipy.spatial as sp, scipy.cluster.hierarchy as hc
flights = sns.load_dataset("flights")
flights = flights.pivot("month", "year", "passengers")
row_linkage, col_linkage = (hc.linkage(sp.distance.pdist(x), method='average')
for x in (flights.values, flights.values.T))
g = sns.clustermap(flights, row_linkage=row_linkage, col_linkage=col_linkage)
# note: this produces the same plot as "sns.clustermap(flights)", where
# clustermap() calculates the row and column linkages internally