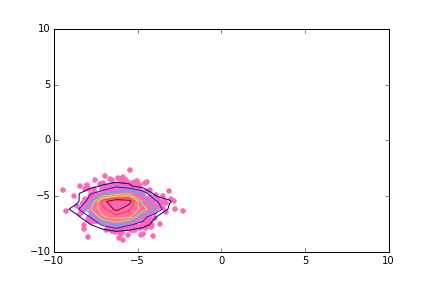
Как нарисовать диаграмму рассеяния с линиями плотности контура в полярных координатах с помощью Matplotlib?
Я пытаюсь сделать точечную диаграмму в полярные координаты с контурными линиями, наложенными на облако точек. Я знаю, как это сделать в декартовых координатах, используя numpy.histogram2d:
# Simple case: scatter plot with density contours in cartesian coordinates
import matplotlib.pyplot as pl
import numpy as np
np.random.seed(2015)
N = 1000
shift_value = -6.
x1 = np.random.randn(N) + shift_value
y1 = np.random.randn(N) + shift_value
fig, ax = pl.subplots(nrows=1,ncols=1)
ax.scatter(x1,y1,color='hotpink')
H, xedges, yedges = np.histogram2d(x1,y1)
extent = [xedges[0],xedges[-1],yedges[0],yedges[-1]]
cset1 = ax.contour(H,extent=extent)
# Modify xlim and ylim to be a bit more consistent with what's next
ax.set_xlim(xmin=-10.,xmax=+10.)
ax.set_ylim(ymin=-10.,ymax=+10.)
выход здесь:
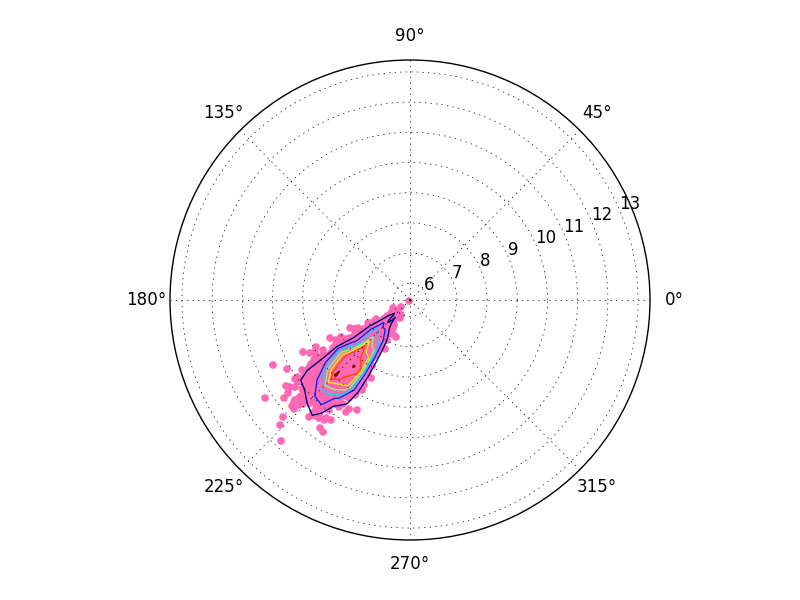
однако, когда я пытаюсь перенести свой код на полярные координаты, я получаю искаженные контурные линии. Вот мой код и произведенный (неправильный) вывод:
# Case with polar coordinates; the contour lines are distorted
np.random.seed(2015)
N = 1000
shift_value = -6.
def CartesianToPolar(x,y):
r = np.sqrt(x**2 + y**2)
theta = np.arctan2(y,x)
return theta, r
x2 = np.random.randn(N) + shift_value
y2 = np.random.randn(N) + shift_value
theta2, r2 = CartesianToPolar(x2,y2)
fig2 = pl.figure()
ax2 = pl.subplot(projection="polar")
ax2.scatter(theta2, r2, color='hotpink')
H, xedges, yedges = np.histogram2d(x2,y2)
theta_edges, r_edges = CartesianToPolar(xedges[:-1],yedges[:-1])
ax2.contour(theta_edges, r_edges,H)
на неправильно вывод здесь:
есть ли способ иметь контурные линии в правильном масштабе?
EDIT для рассмотрения предложений, сделанных в комментариях.
EDIT2: кто-то предположил, что вопрос может быть дубликатом этот вопрос. Хотя я признаю, что проблемы схожи, моя касается конкретно построения контуров плотности точек над диаграммой рассеяния. Другой вопрос о том, как построить контур уровни любого количества, заданного вместе с координатами точек.
1 ответов
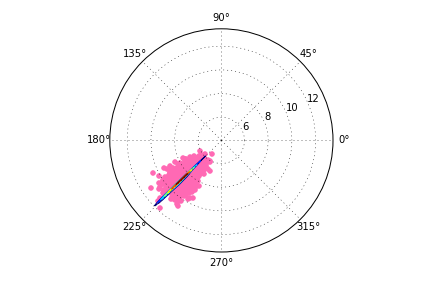
проблема в том, что вы конвертируете только края массива. Преобразуя только координаты X и y ребер, вы эффективно преобразуете координаты диагональной линии через 2D-массив. Эта линия имеет очень маленький диапазон theta значения, и вы применяете этот диапазон ко всей сетке.
быстрое (но неправильное) исправление
в большинстве случаев вы можете преобразовать всю сетку (т. е. 2D-массивы x и y, изготовления 2D массивы theta и r) в полярных координатах.
вместо:
H, xedges, yedges = np.histogram2d(x2,y2)
theta_edges, r_edges = CartesianToPolar(xedges[:-1],yedges[:-1])
сделать что-то похожее на:
H, xedges, yedges = np.histogram2d(x2,y2)
xedges, yedges = np.meshgrid(xedges[:-1],yedges[:-1]
theta_edges, r_edges = CartesianToPolar(xedges, yedges)
как пример:
import numpy as np
import matplotlib.pyplot as plt
def main():
x2, y2 = generate_data()
theta2, r2 = cart2polar(x2,y2)
fig2 = plt.figure()
ax2 = fig2.add_subplot(111, projection="polar")
ax2.scatter(theta2, r2, color='hotpink')
H, xedges, yedges = np.histogram2d(x2,y2)
xedges, yedges = np.meshgrid(xedges[:-1], yedges[:-1])
theta_edges, r_edges = cart2polar(xedges, yedges)
ax2.contour(theta_edges, r_edges, H)
plt.show()
def generate_data():
np.random.seed(2015)
N = 1000
shift_value = -6.
x2 = np.random.randn(N) + shift_value
y2 = np.random.randn(N) + shift_value
return x2, y2
def cart2polar(x,y):
r = np.sqrt(x**2 + y**2)
theta = np.arctan2(y,x)
return theta, r
main()
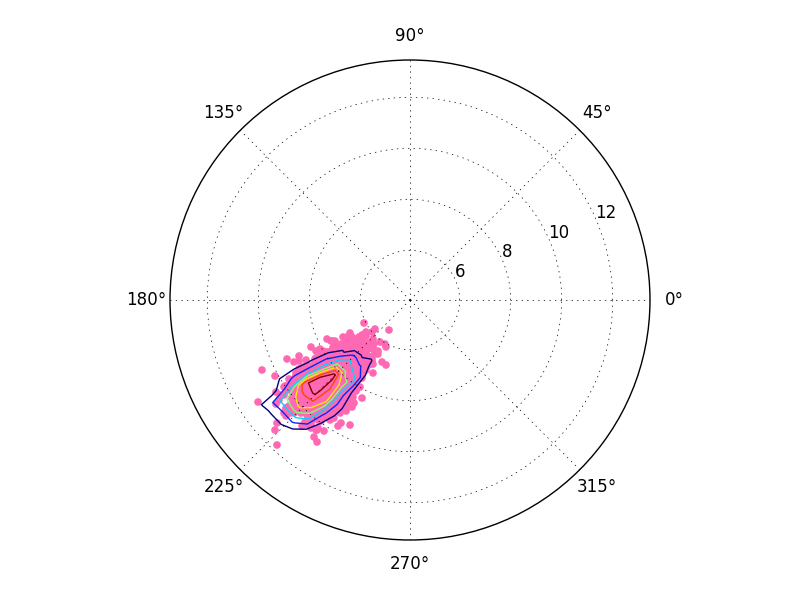
ax.contour неявно предполагает, что входные данные на регулярной сетке. Мы дали ему регулярную сетку в декартовых координатах, но не регулярную сетку в полярных координирует. Предполагается, что мы прошли через обычную сетку в полярных координатах. Мы могли бы перепроверить сетку, но есть более простой способ.
правильное решение
чтобы правильно построить 2D-гистограмму, вычислите гистограмму в Полярном пространстве.
например, сделать что-то похожее на:
theta2, r2 = cart2polar(x2,y2)
H, theta_edges, r_edges = np.histogram2d(theta2, r2)
ax2.contour(theta_edges[:-1], r_edges[:-1], H)
как пример:
import numpy as np
import matplotlib.pyplot as plt
def main():
x2, y2 = generate_data()
theta2, r2 = cart2polar(x2,y2)
fig2 = plt.figure()
ax2 = fig2.add_subplot(111, projection="polar")
ax2.scatter(theta2, r2, color='hotpink')
H, theta_edges, r_edges = np.histogram2d(theta2, r2)
ax2.contour(theta_edges[:-1], r_edges[:-1], H)
plt.show()
def generate_data():
np.random.seed(2015)
N = 1000
shift_value = -6.
x2 = np.random.randn(N) + shift_value
y2 = np.random.randn(N) + shift_value
return x2, y2
def cart2polar(x,y):
r = np.sqrt(x**2 + y**2)
theta = np.arctan2(y,x)
return theta, r
main()
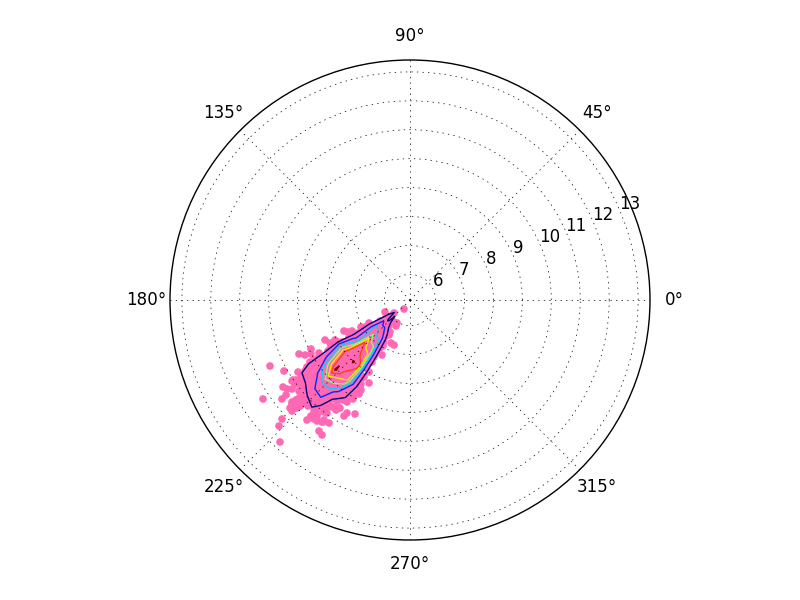
наконец, вы можете заметить небольшое изменение в приведенном выше результате. Это связано с соглашениями сетки, ориентированными на ячейки (x[0,0], y[0,0] дает центр ячейки) против ориентированных на ребро соглашений сетки (x[0,0], y[0,0] дает нижний левый угол ячейки. ax.contour ожидает, что все будет центрировано по ячейкам, но вы даете ему выровненные по краю значения x и y.
это всего лишь полуклеточный сдвиг, но если вы хотите его исправить, Сделайте что-то вроде:
def centers(bins):
return np.vstack([bins[:-1], bins[1:]]).mean(axis=0)
H, theta_edges, r_edges = np.histogram2d(theta2, r2)
theta_centers, r_centers = centers(theta_edges), centers(r_edges)
ax2.contour(theta_centers, r_centers, H)