как я могу получить поддеревья дендрограммы, сделанные scipy.группа.иерархия
у меня была путаница в отношении этого модуля (scipy.группа.иерархия. ).. и все еще есть !
например, у нас есть следующая дендрограмма:
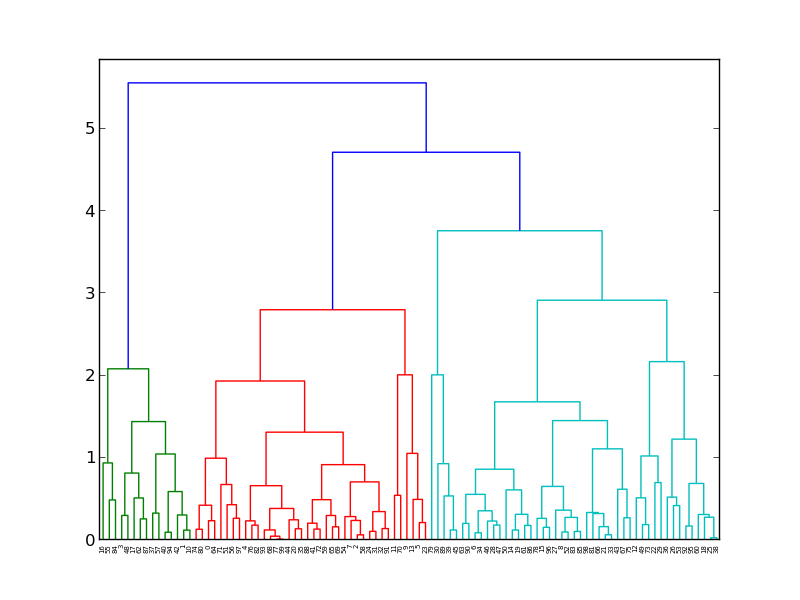
мой вопрос в том, как я могу извлечь цветные поддеревья (каждый из которых представляет кластер) в хорошем формате, скажем, в формате SIF ? Теперь код, чтобы получить сюжет выше:
import scipy
import scipy.cluster.hierarchy as sch
import matplotlib.pylab as plt
scipy.randn(100,2)
d = sch.distance.pdist(X)
Z= sch.linkage(d,method='complete')
P =sch.dendrogram(Z)
plt.savefig('plot_dendrogram.png')
T = sch.fcluster(Z, 0.5*d.max(), 'distance')
#array([4, 5, 3, 2, 2, 3, 5, 2, 2, 5, 2, 2, 2, 3, 2, 3, 2, 5, 4, 5, 2, 5, 2,
# 3, 3, 3, 1, 3, 4, 2, 2, 4, 2, 4, 3, 3, 2, 5, 5, 5, 3, 2, 2, 2, 5, 4,
# 2, 4, 2, 2, 5, 5, 1, 2, 3, 2, 2, 5, 4, 2, 5, 4, 3, 5, 4, 4, 2, 2, 2,
# 4, 2, 5, 2, 2, 3, 3, 2, 4, 5, 3, 4, 4, 2, 1, 5, 4, 2, 2, 5, 5, 2, 2,
# 5, 5, 5, 4, 3, 3, 2, 4], dtype=int32)
sch.leaders(Z,T)
# (array([190, 191, 182, 193, 194], dtype=int32),
# array([2, 3, 1, 4,5],dtype=int32))
Итак, вывод fcluster() дает кластеризацию узлов (по их идентификаторам) и leaders() описал здесь предполагается вернуть 2 массива:
первый содержит ведущие узлы кластеров, генерируемых Z, здесь мы видим, что у нас есть 5 кластеров, а также на графике
и второй идентификатор этих кластеров
Итак, если этот leaders () возвращает resp. L и M:L[2]=182 и M[2]=1, затем кластер 1 возглавляется узлом id 182, который не существует в наборе наблюдений X, в документации сказано "... тогда это соответствует не-синглетному кластеру". Но я не могу его достать ...
кроме того, я преобразовал Z в дерево с помощью sch.to_tree(Z), это вернет простой в использовании объект дерева, который я хочу визуализировать, но какой инструмент я должен использовать в качестве графической платформы, которая управляет этими объектами дерева в качестве входных данных?
1 ответов
отвечая на часть вашего вопроса о манипуляции с деревом...
как поясняется в адругие ответ, вы можете прочитать координаты филиалов чтение icoord и dcoord из дерева объектов. Для каждой ветви координаты даются слева направо.
если вы хотите построить вручную дерево, вы можете использовать что-то вроде:
def plot_tree( P, pos=None ):
icoord = scipy.array( P['icoord'] )
dcoord = scipy.array( P['dcoord'] )
color_list = scipy.array( P['color_list'] )
xmin, xmax = icoord.min(), icoord.max()
ymin, ymax = dcoord.min(), dcoord.max()
if pos:
icoord = icoord[pos]
dcoord = dcoord[pos]
color_list = color_list[pos]
for xs, ys, color in zip(icoord, dcoord, color_list):
plt.plot(xs, ys, color)
plt.xlim( xmin-10, xmax + 0.1*abs(xmax) )
plt.ylim( ymin, ymax + 0.1*abs(ymax) )
plt.show()
где, в коде plot_tree(P) дает:
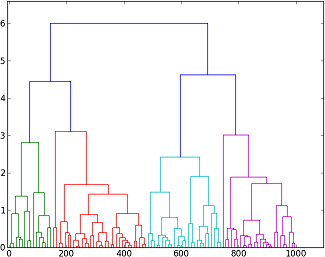
функция позволяет выбрать только некоторые отрасли:
plot_tree(P, range(10))
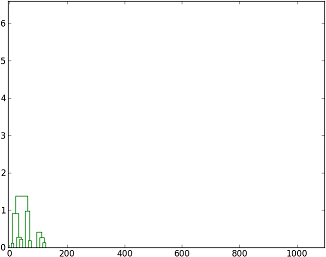
теперь вы должны знать, какие ветви строить. Может быть,fcluster() выход немного неясен и другой способ найти, какие ветви для построения на основе минимального и максимального допуска расстояния будут использовать выход linkage() напрямую (Z в случае OP):
dmin = 0.2
dmax = 0.3
pos = scipy.all( (Z[:,2] >= dmin, Z[:,2] <= dmax), axis=0 ).nonzero()
plot_tree( P, pos )
рекомендовано литература:
