Линейная пригонка включая все ошибки с NumPy/SciPy
у меня есть много точек данных x-y с ошибками на y, которые мне нужно соответствовать нелинейным функциям. Эти функции могут быть линейными в некоторых случаях, но чаще являются экспоненциальным распадом, кривыми Гаусса и т. д. SciPy поддерживает этот вид подгонки с scipy.optimize.curve_fit, и я также могу указать вес каждой точки. Это дает мне взвешенный нелинейный фитинг, который великолепен. Из результатов я могу извлечь параметры и их соответствующие ошибки.
есть только одно предостережение: ошибки используются только в качестве весов, но не включены в ошибку. Если я удвою ошибки во всех моих точках данных, я ожидаю, что неопределенность результата также увеличится. Поэтому я построил тестовый пример (исходный код), чтобы проверить это.
Fit с scipy.optimize.curve_fit дает мне:
Parameters: [ 1.99900756 2.99695535]
Errors: [ 0.00424833 0.00943236]
то же самое, но с 2 * y_err:
Parameters: [ 1.99900756 2.99695535]
Errors: [ 0.00424833 0.00943236]
то же самое, но с 2 * y_err:
таким образом, вы можете видеть, что значения идентичны. Это говорит мне, что алгоритм не учитывает их, но я думаю, что значения должны быть разными.
Я читал о другом методе подгонки здесь, поэтому я попытался соответствовать scipy.odr а также:
Beta: [ 2.00538124 2.95000413]
Beta Std Error: [ 0.00652719 0.03870884]
то же самое, но с 20 * y_err:
Beta: [ 2.00517894 2.9489472 ]
Beta Std Error: [ 0.00642428 0.03647149]
значения немного отличаются, но я думаю, что это объясняет увеличение ошибки на всех. Я думаю, что это просто ошибки округления или немного разных весовых.
есть ли какой-то пакет это позволяет мне соответствовать данным и получать фактические ошибки? У меня есть формулы здесь, в книге, но я не хочу реализовывать это сам, если мне не нужно.
теперь я прочитал о linfit.py другой вопрос. Это очень хорошо справляется с тем, что я имею в виду. Он поддерживает оба режима, и первый-это то, что мне нужно.
Fit with linfit:
Parameters: [ 2.02600849 2.91759066]
Errors: [ 0.00772283 0.04449971]
Same but with 20 * y_err:
Parameters: [ 2.02600849 2.91759066]
Errors: [ 0.15445662 0.88999413]
Fit with linfit(relsigma=True):
Parameters: [ 2.02600849 2.91759066]
Errors: [ 0.00622595 0.03587451]
Same but with 20 * y_err:
Parameters: [ 2.02600849 2.91759066]
Errors: [ 0.00622595 0.03587451]
должен ли я ответить на свой вопрос или просто закрыть/удалить его сейчас?
3 ответов
Пожалуйста, обратите внимание, что из документации curvefit:
Сигма: нет или N-длина последовательности Если нет, этот вектор будет использоваться в качестве относительного веса в проблема наименьших квадратов.
ключевым моментом здесь является как относительные веса, поэтому yerr в строке 53 и 2*yerr в 57 должен дать вам аналогичный, если не тот же результат.
при увеличении на самом деле ошибка остатков , вы увидите, что значения в ковариационной матрице увеличиваются. Скажите, если мы изменим y += random to y += 5*random функции generate_data():
Fit with scipy.optimize.curve_fit:
('Parameters:', array([ 1.92810458, 3.97843448]))
('Errors: ', array([ 0.09617346, 0.64127574]))
сравнивает с первоначальным результатом:
Fit with scipy.optimize.curve_fit:
('Parameters:', array([ 2.00760386, 2.97817514]))
('Errors: ', array([ 0.00782591, 0.02983339]))
также обратите внимание, что оценка параметра теперь дальше от (2,3), как и следовало ожидать от увеличенной ошибки остатка и большего доверительного интервала оценок параметров.
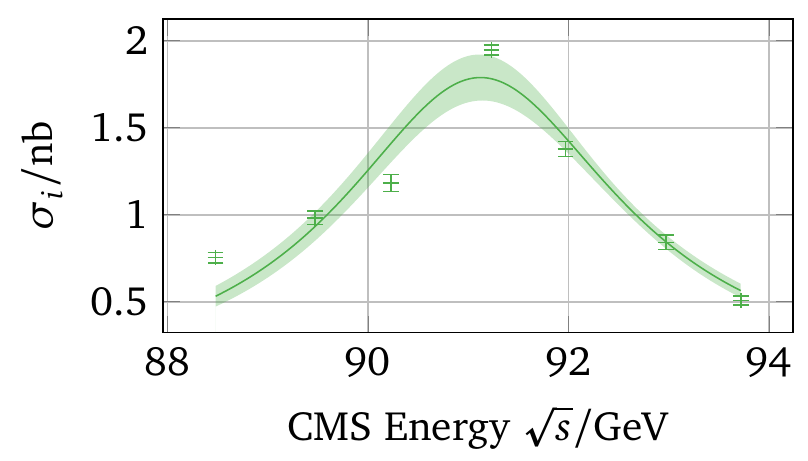
один из способов, который работает хорошо и на самом деле дает лучший результат, - это метод bootstrap. Когда даны точки данных с ошибками, используется параметрический бутстрап и пусть каждый x и y значение опишите гауссово распределение. Затем один из них нарисует точку из каждого из этих дистрибутивов и получит новый загрузочный образец. Выполнение простой невзвешенной подгонки дает одно значение для параметров.
этот процесс повторяется от 300 до нескольких тысяч раз. Одна воля в конечном итоге с распределением параметров подгонки, где можно взять среднее и стандартное отклонение для получения значения и ошибки.
еще одна аккуратная вещь заключается в том, что в результате не получается ни одной кривой подгонки, но их много. Для каждого интерполированного x значение можно снова взять среднее и стандартное отклонение многих значений f(x, param) и получить диапазон ошибок:
дальнейшие шаги в анализе затем снова выполняется сотни раз с различными параметрами подгонки. Это также будет учитывать корреляцию параметров подгонки, как ясно видно на графике выше: хотя симметричная функция была установлена на данные, полоса ошибок асимметрична. Это будет означать, что интерполированные значения слева имеют большую неопределенность, чем справа.
короткий ответ:
для абсолютных значений, которые включают неопределенность в y (и в X для случая odr):
- на
scipy.odrcase usestddev = numpy.sqrt(numpy.diag(cov))где cov - ковариационная матрица, которую odr дает на выходе. - на
scipy.optimize.curve_fitпример использованияabsolute_sigma=True
флаг.
для относительных значений (исключая неопределенность):
на
scipy.odrслучае используйте значение sd из выходных данных.В составляющей.оптимизировать.curve_fit case use
absolute_sigma=Falseфлаг.используйте numpy.polyfit такой:
p, cov = numpy.polyfit(x, y, 1,cov = True)
errorbars = numpy.sqrt(numpy.diag(cov))
ответ
существует некоторое недокументированное поведение во всех функциях. Я предполагаю, что функции смешивают относительные и абсолютные значения. В конце этот ответ-это код, который либо дает то, что вы хотите (или нет) на основе того, как вы обрабатываете вывод (есть ошибка?). Кроме того, curve_fit, возможно, недавно получил флаг "absolute_sigma"?
моя точка находится в выходе. Кажется, что odr вычисляет стандартное отклонение, поскольку нет неопределенностей, подобных polyfit, но если стандартное отклонение вычисляется из ковариационной матрицы, неопределенности существуют. Curve_fit делает это с absolute_sigma=True флаг. Ниже приведен вывод, содержащий
- диагональные элементы ковариационной матрицы cov (0,0) и
- ков(1,1),
- неправильно путь для стандартного отклонения от выходов для наклона и
- неправильно путь для постоянного и
- право путь для стандартного отклонения от выходов для наклона и
- право путь к константа
odr: 1.739631e-06 0.02302262 [ 0.00014863 0.0170987 ] [ 0.00131895 0.15173207]
curve_fit: 2.209469e-08 0.00029239 [ 0.00014864 0.01709943] [ 0.0004899 0.05635713]
polyfit: 2.232016e-08 0.00029537 [ 0.0001494 0.01718643]
обратите внимание, что odr и polyfit имеют точно такое же стандартное отклонение. Polyfit делает не получаем неопределенность как вход так odr не использует неопределенности при расчете стандартного отклонения. Ковариационная матрица использует их, и если в случае odr стандартное отклонение вычисляется из неопределенности ковариационной матрицы, они изменяются при увеличении неопределенности. Возиться с dy в коде ниже покажет это.
я пишу это здесь в основном потому, что это важно знать при поиске пределов ошибок (и руководство fortran odrpack, куда ссылается scipy, имеет некоторую вводящую в заблуждение информацию об этом: стандартное отклонение должно быть квадратным корнем ковариационной матрицы, как говорит руководство, но это не так).
import scipy.odr
import scipy.optimize
import numpy
x = numpy.arange(200)
y = x + 0.4*numpy.random.random(x.shape)
dy = 0.4
def stddev(cov): return numpy.sqrt(numpy.diag(cov))
def f(B, x): return B[0]*x + B[1]
linear = scipy.odr.Model(f)
mydata = scipy.odr.RealData(x, y, sy = dy)
myodr = scipy.odr.ODR(mydata, linear, beta0 = [1.0, 1.0], sstol = 1e-20, job=00000)
myoutput = myodr.run()
cov = myoutput.cov_beta
sd = myoutput.sd_beta
p = myoutput.beta
print 'odr: ', cov[0,0], cov[1,1], sd, stddev(cov)
p2, cov2 = scipy.optimize.curve_fit(lambda x, a, b:a*x+b,
x, y, [1,1],
sigma = dy,
absolute_sigma = False,
xtol = 1e-20)
p3, cov3 = scipy.optimize.curve_fit(lambda x, a, b:a*x+b,
x, y, [1,1],
sigma = dy,
absolute_sigma = True,
xtol = 1e-20)
print 'curve_fit: ', cov2[0,0], cov2[1,1], stddev(cov2), stddev(cov3)
p, cov4 = numpy.polyfit(x, y, 1,cov = True)
print 'polyfit: ', cov4[0,0], cov4[1,1], stddev(cov4)