Networkx: извлечение связанного компонента, содержащего данный узел (направленный граф)
Я пытаюсь извлечь из большого графика подграф всех связанных узлов, содержащих определенный узел.
есть ли решение в библиотеке Networkx?
[EDIT]
Мой график-это Орграф
[EDIT]
Перефразировал просто:
Я хочу, чтобы часть моего графика, который содержит мой конкретный узел N_i и все узлы, которые связаны прямо или косвенно (проходя мимо других узлов), используя любые входящие или исходящие стыки.
Пример:
>>> g = nx.DiGraph()
>>> g.add_path(['A','B','C',])
>>> g.add_path(['X','Y','Z',])
>>> g.edges()
[('A', 'B'), ('B', 'C'), ('Y', 'Z'), ('X', 'Y')]
мой желаемый результат будет:
>>> g2 = getSubGraph(g, 'B')
>>> g2.nodes()
['A', 'B', 'C']
>>> g2.edges()
[('A', 'B'), ('B', 'C')]
4 ответов
вы можете использовать shortest_path (), чтобы найти все узлы, доступные из данного узла. В вашем случае вам нужно сначала преобразовать график в неориентированное представление, чтобы следовать как в -, так и за-ребрами.
In [1]: import networkx as nx
In [2]: >>> g = nx.DiGraph()
In [3]: >>> g.add_path(['A','B','C',])
In [4]: >>> g.add_path(['X','Y','Z',])
In [5]: u = g.to_undirected()
In [6]: nodes = nx.shortest_path(u,'B').keys()
In [7]: nodes
Out[7]: ['A', 'C', 'B']
In [8]: s = g.subgraph(nodes)
In [9]: s.edges()
Out[9]: [('A', 'B'), ('B', 'C')]
или в одной строке
In [10]: s = g.subgraph(nx.shortest_path(g.to_undirected(),'B'))
In [11]: s.edges()
Out[11]: [('A', 'B'), ('B', 'C')]
просто выполните цикл через подграфы, пока целевой узел не будет содержаться в подграфе.
на направлено графики, я предполагаю, что подграф-это граф, такой, что каждый узел доступен из каждого другого узла. Это сильно связный подграфа и
используйте пример в конце страницы connected_component_subgraphs.
просто убедитесь, что ссылаются на последний элемент из списка, а не на первый
>>> G=nx.path_graph(4)
>>> G.add_edge(5,6)
>>> H=nx.connected_component_subgraphs(G)[-1]
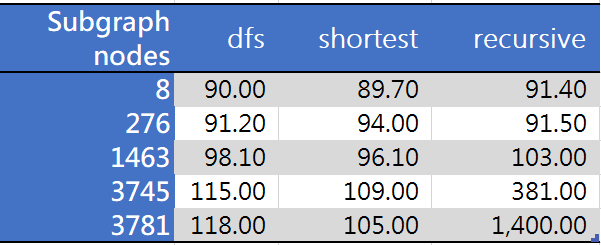
Я нашел три решения для решения вашего требования, так же, как и мое. Размер моего Орграфа составляет от 6000 до 12000 узлов, а максимальный размер подграфа-до 3700. Три функции, которые я использовал:
def create_subgraph_dfs(G, node):
""" bidirection, O(1)"""
edges = nx.dfs_successors(G, node)
nodes = []
for k,v in edges.items():
nodes.extend([k])
nodes.extend(v)
return G.subgraph(nodes)
def create_subgraph_shortpath(G, node):
""" unidirection, O(1)"""
nodes = nx.single_source_shortest_path(G,node).keys()
return G.subgraph(nodes)
def create_subgraph_recursive(G, sub_G, start_node):
""" bidirection, O(nlogn)"""
for n in G.successors_iter(start_node):
sub_G.add_path([start_node, n])
create_subgraph_recursive(G, sub_G, n)
результат теста сводный следующим образом: