Поместите звезды на ggplot barplots и boxplots - чтобы указать уровень значимости (p-значение)
обычно звезды помещаются на барплоты или boxplots, чтобы показать уровень значимости (p-value) одной или двух групп, ниже приведены несколько примеров:
число звезд определяется p-значением, например, можно поставить 3 звезды для p-значения
и мои вопросы: Как генерировать подобные диаграммы? Методы, которые автоматически помещенные звезды, основанные на уровне значимости, более чем приветствуются.
4 ответов
пожалуйста, найдите мою попытку ниже.
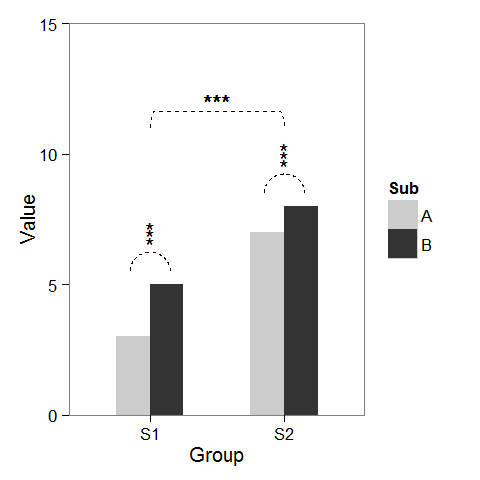
во-первых, я создал некоторые фиктивные данные и barplot, которые могут быть изменены по нашему желанию.
windows(4,4)
dat <- data.frame(Group = c("S1", "S1", "S2", "S2"),
Sub = c("A", "B", "A", "B"),
Value = c(3,5,7,8))
## Define base plot
p <-
ggplot(dat, aes(Group, Value)) +
theme_bw() + theme(panel.grid = element_blank()) +
coord_cartesian(ylim = c(0, 15)) +
scale_fill_manual(values = c("grey80", "grey20")) +
geom_bar(aes(fill = Sub), stat="identity", position="dodge", width=.5)
добавление звездочек над столбцом легко, как уже упоминал Батист. Просто создайте data.frame с координатами.
label.df <- data.frame(Group = c("S1", "S2"),
Value = c(6, 9))
p + geom_text(data = label.df, label = "***")
чтобы добавить дуги, указывающие на сравнение подгрупп, я вычислил параметрические координаты полукруга и добавил их, связанные с geom_line. Звездочки нужны новые координаты тоже.
label.df <- data.frame(Group = c(1,1,1, 2,2,2),
Value = c(6.5,6.8,7.1, 9.5,9.8,10.1))
# Define arc coordinates
r <- 0.15
t <- seq(0, 180, by = 1) * pi / 180
x <- r * cos(t)
y <- r*5 * sin(t)
arc.df <- data.frame(Group = x, Value = y)
p2 <-
p + geom_text(data = label.df, label = "*") +
geom_line(data = arc.df, aes(Group+1, Value+5.5), lty = 2) +
geom_line(data = arc.df, aes(Group+2, Value+8.5), lty = 2)
наконец, чтобы указать сравнение между группами, я построил больший круг и сплющил его вверху.
r <- .5
x <- r * cos(t)
y <- r*4 * sin(t)
y[20:162] <- y[20] # Flattens the arc
arc.df <- data.frame(Group = x, Value = y)
p2 + geom_line(data = arc.df, aes(Group+1.5, Value+11), lty = 2) +
geom_text(x = 1.5, y = 12, label = "***")
Я знаю, что это старый вопрос, и ответ Йенса Тирлинга уже дает одно решение проблемы. Но недавно я создал ggplot-расширение, которое упрощает весь процесс добавления баров значимости:ggsignif
вместо того, чтобы нудно добавлять geom_line и geom_text к вашему сюжету вы просто добавляете один слой geom_signif:
library(ggplot2)
library(ggsignif)
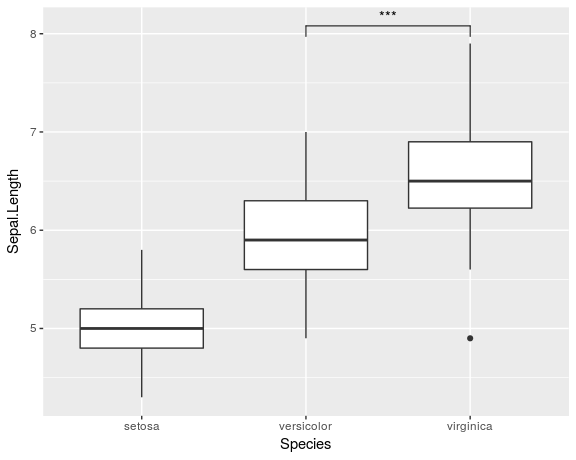
ggplot(iris, aes(x=Species, y=Sepal.Length)) +
geom_boxplot() +
geom_signif(comparisons = list(c("versicolor", "virginica")),
map_signif_level=TRUE)
чтобы создать более продвинутый сюжет, похожий на тот показано Йенс Tierling, вы можете сделать:
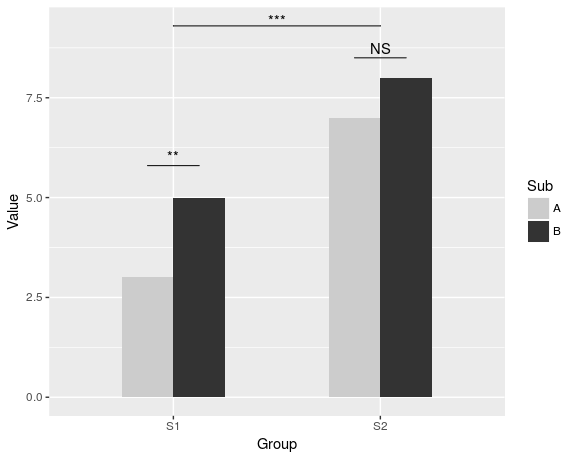
dat <- data.frame(Group = c("S1", "S1", "S2", "S2"),
Sub = c("A", "B", "A", "B"),
Value = c(3,5,7,8))
ggplot(dat, aes(Group, Value)) +
geom_bar(aes(fill = Sub), stat="identity", position="dodge", width=.5) +
geom_signif(stat="identity",
data=data.frame(x=c(0.875, 1.875), xend=c(1.125, 2.125),
y=c(5.8, 8.5), annotation=c("**", "NS")),
aes(x=x,xend=xend, y=y, yend=y, annotation=annotation)) +
geom_signif(comparisons=list(c("S1", "S2")), annotations="***",
y_position = 9.3, tip_length = 0, vjust=0.4) +
scale_fill_manual(values = c("grey80", "grey20"))
полная документация пакета доступна по адресу кран.
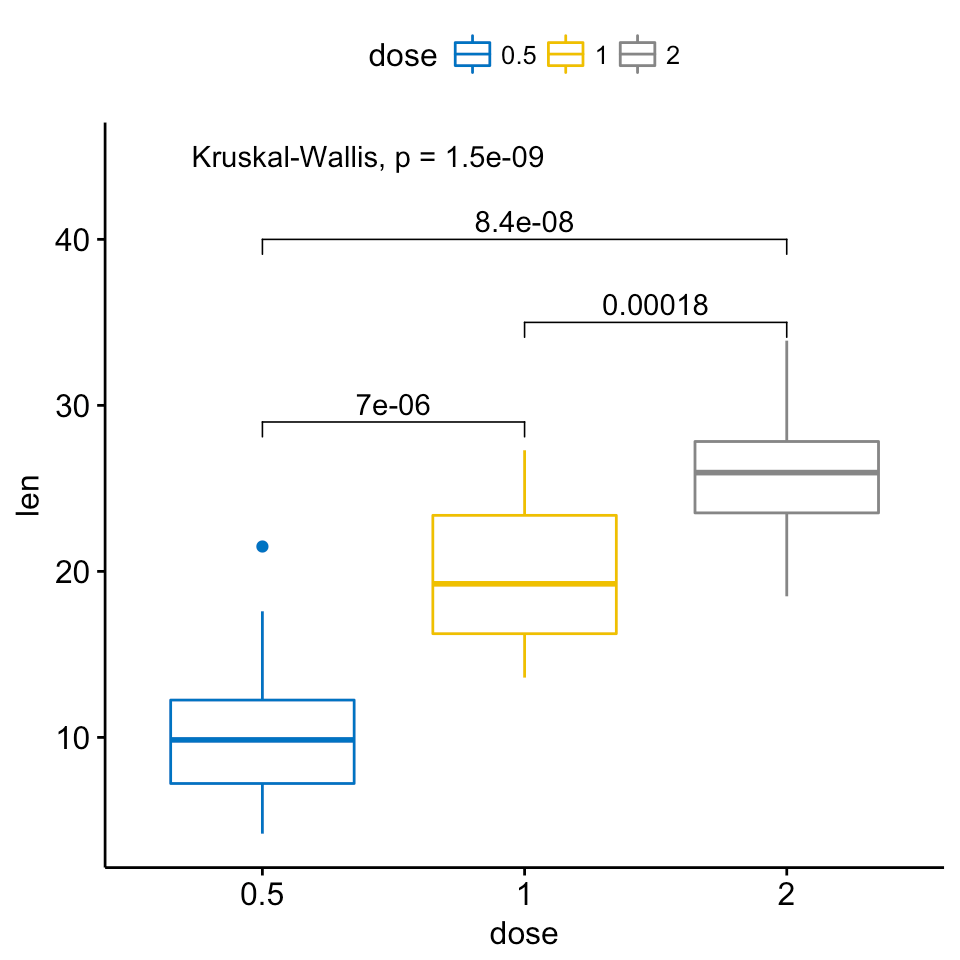
существует также расширение ggsignif под названием ggpubr это более мощный, когда дело доходит до сравнения нескольких групп. Он строится поверх ggsignif, но также обрабатывает anova и kruskal-wallis, а также попарные сравнения с гобальным средним.
пример:
ggboxplot(ToothGrowth, x = "dose", y = "len",
color = "dose", palette = "jco")+
stat_compare_means(comparisons = my_comparisons, label.y = c(29, 35, 40))+
stat_compare_means(label.y = 45)
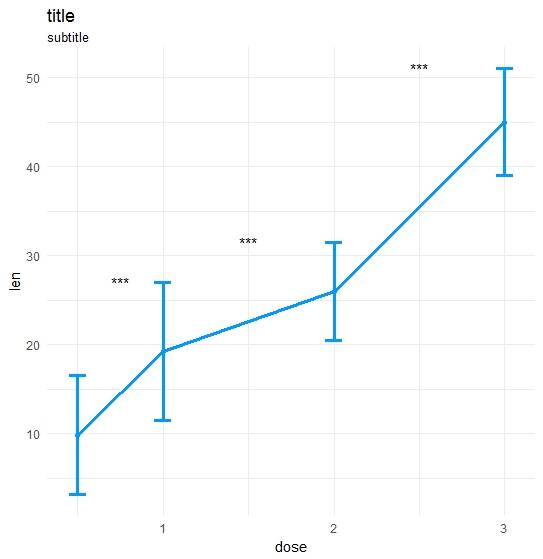
сделал свою собственную функцию:
ts_test <- function(dataL,x,y,method="t.test",idCol=NULL,paired=F,label = "p.signif",p.adjust.method="none",alternative = c("two.sided", "less", "greater"),...) {
options(scipen = 999)
annoList <- list()
setDT(dataL)
if(paired) {
allSubs <- dataL[,.SD,.SDcols=idCol] %>% na.omit %>% unique
dataL <- dataL[,merge(.SD,allSubs,by=idCol,all=T),by=x] #idCol!!!
}
if(method =="t.test") {
dataA <- eval(parse(text=paste0(
"dataL[,.(",as.name(y),"=mean(get(y),na.rm=T),sd=sd(get(y),na.rm=T)),by=x] %>% setDF"
)))
res<-pairwise.t.test(x=dataL[[y]], g=dataL[[x]], p.adjust.method = p.adjust.method,
pool.sd = !paired, paired = paired,
alternative = alternative, ...)
}
if(method =="wilcox.test") {
dataA <- eval(parse(text=paste0(
"dataL[,.(",as.name(y),"=median(get(y),na.rm=T),sd=IQR(get(y),na.rm=T,type=6)),by=x] %>% setDF"
)))
res<-pairwise.wilcox.test(x=dataL[[y]], g=dataL[[x]], p.adjust.method = p.adjust.method,
paired = paired, ...)
}
#Output the groups
res$p.value %>% dimnames %>% {paste(.[[2]],.[[1]],sep="_")} %>% cat("Groups ",.)
#Make annotations ready
annoList[["label"]] <- res$p.value %>% diag %>% round(5)
if(!is.null(label)) {
if(label == "p.signif"){
annoList[["label"]] %<>% cut(.,breaks = c(-0.1, 0.0001, 0.001, 0.01, 0.05, 1),
labels = c("****", "***", "**", "*", "ns")) %>% as.character
}
}
annoList[["x"]] <- dataA[[x]] %>% {diff(.)/2 + .[-length(.)]}
annoList[["y"]] <- {dataA[[y]] + dataA[["sd"]]} %>% {pmax(lag(.), .)} %>% na.omit
#Make plot
coli="#0099ff";sizei=1.3
p <-ggplot(dataA, aes(x=get(x), y=get(y))) +
geom_errorbar(aes(ymin=len-sd, ymax=len+sd),width=.1,color=coli,size=sizei) +
geom_line(color=coli,size=sizei) + geom_point(color=coli,size=sizei) +
scale_color_brewer(palette="Paired") + theme_minimal() +
xlab(x) + ylab(y) + ggtitle("title","subtitle")
#Annotate significances
p <-p + annotate("text", x = annoList[["x"]], y = annoList[["y"]], label = annoList[["label"]])
return(p)
}
Data и звонок:
library(ggplot2);library(data.table);library(magrittr);
df_long <- rbind(ToothGrowth[,-2],data.frame(len=40:50,dose=3.0))
df_long$ID <- data.table::rowid(df_long$dose)
ts_test(dataL=df_long,x="dose",y="len",idCol="ID",method="wilcox.test",paired=T)