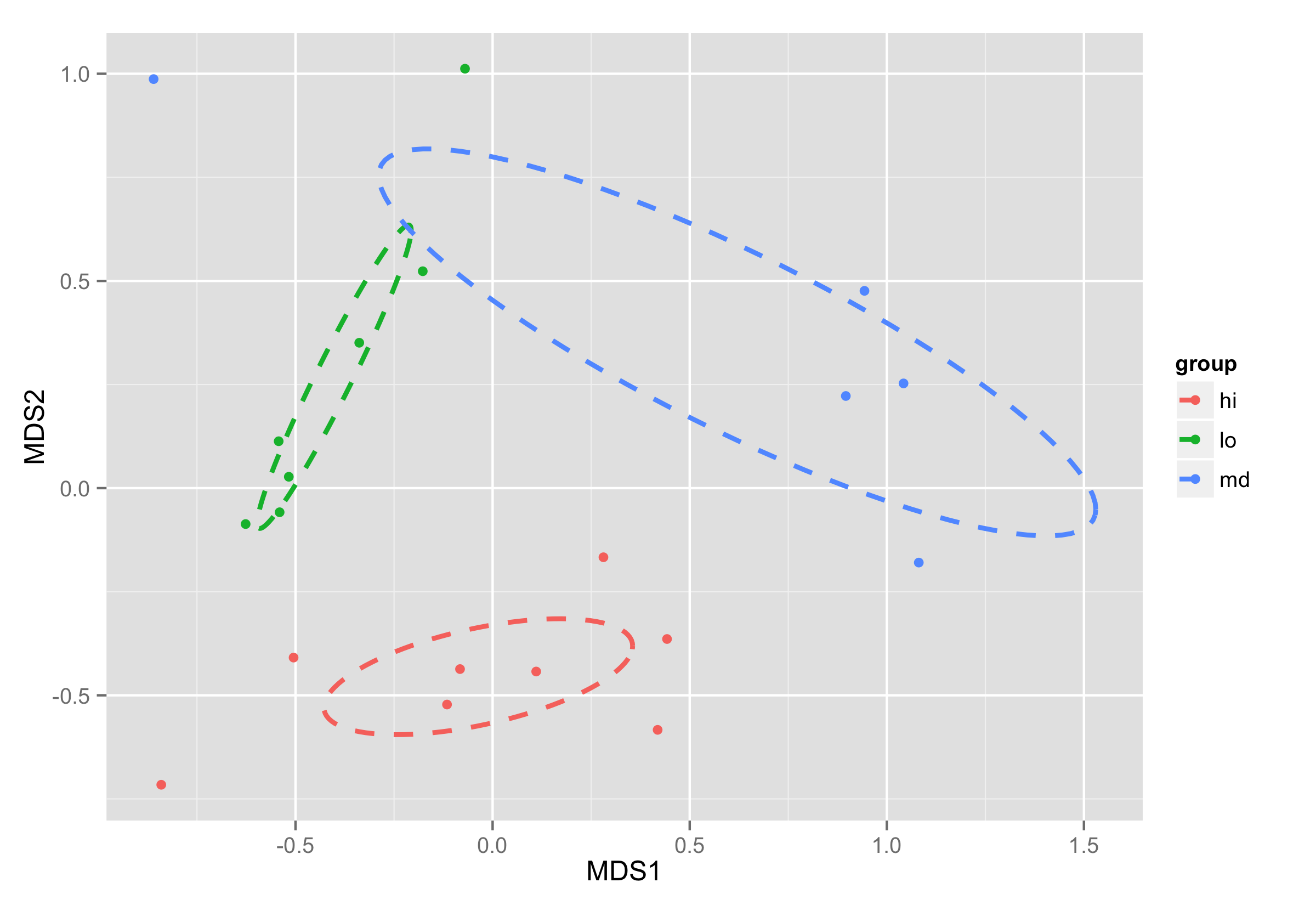
Построение функции ordiellipse из пакета vegan на график NMDS, созданный в ggplot2
вместо нормальной функции графика я использую ggplot2 для создания графиков NMDS. Я хотел бы отобразить группы на графике NMDS, используя функцию ordiellipse() С .
пример:
library(vegan)
library(ggplot2)
data(dune)
# calculate distance for NMDS
sol <- metaMDS(dune)
# Create meta data for grouping
MyMeta = data.frame(
sites = c(2,13,4,16,6,1,8,5,17,15,10,11,9,18,3,20,14,19,12,7),
amt = c("hi", "hi", "hi", "md", "lo", "hi", "hi", "lo", "md", "md", "lo",
"lo", "hi", "lo", "hi", "md", "md", "lo", "hi", "lo"),
row.names = "sites")
# plot NMDS using basic plot function and color points by "amt" from MyMeta
plot(sol$points, col = MyMeta$amt)
# draw dispersion ellipses around data points
ordiellipse(sol, MyMeta$amt, display = "sites", kind = "sd", label = T)
# same in ggplot2
NMDS = data.frame(MDS1 = sol$points[,1], MDS2 = sol$points[,2])
ggplot(data = NMDS, aes(MDS1, MDS2)) +
geom_point(aes(data = MyMeta, color = MyMeta$amt))
как добавить ordiellipse в график NMDS, созданный с помощью ggplot2?
Элфертс Дидзис' ответ ниже работает отлично. Спасибо! Однако теперь я заинтересован в построении следующего ordiellipse к сюжету NMDS, созданному с помощью ggplot2:
ordiellipse(sol, MyMeta$amt, display = "sites", kind = "se", conf = 0.95, label = T)
к сожалению, я не понимаю, достаточно о том, как veganCovEllipse функция работает, чтобы иметь возможность настроить скрипт самостоятельно.
1 ответов
прежде всего, я добавил группу столбцов в ваш фрейм данных NMDS.
NMDS = data.frame(MDS1 = sol$points[,1], MDS2 = sol$points[,2],group=MyMeta$amt)
второй фрейм данных содержит средние значения MDS1 и MDS2 для каждой группы, и он будет использоваться для отображения имен групп на графике
NMDS.mean=aggregate(NMDS[,1:2],list(group=group),mean)
фрейм данных df_ell содержит значения для отображения многоточий. Он рассчитывается с помощью функции veganCovEllipse который скрыт в vegan пакета. Эта функция применяется к каждому уровню NMDS (группы) и использует также функцию cov.wt для расчета ковариации матрица.
veganCovEllipse<-function (cov, center = c(0, 0), scale = 1, npoints = 100)
{
theta <- (0:npoints) * 2 * pi/npoints
Circle <- cbind(cos(theta), sin(theta))
t(center + scale * t(Circle %*% chol(cov)))
}
df_ell <- data.frame()
for(g in levels(NMDS$group)){
df_ell <- rbind(df_ell, cbind(as.data.frame(with(NMDS[NMDS$group==g,],
veganCovEllipse(cov.wt(cbind(MDS1,MDS2),wt=rep(1/length(MDS1),length(MDS1)))$cov,center=c(mean(MDS1),mean(MDS2)))))
,group=g))
}
теперь эллипсы строятся с помощью функции geom_path() и annotate() используется для построения названия группы.
ggplot(data = NMDS, aes(MDS1, MDS2)) + geom_point(aes(color = group)) +
geom_path(data=df_ell, aes(x=MDS1, y=MDS2,colour=group), size=1, linetype=2)+
annotate("text",x=NMDS.mean$MDS1,y=NMDS.mean$MDS2,label=NMDS.mean$group)
идея построения эллипсов была взята из другого stackoverflow вопрос.
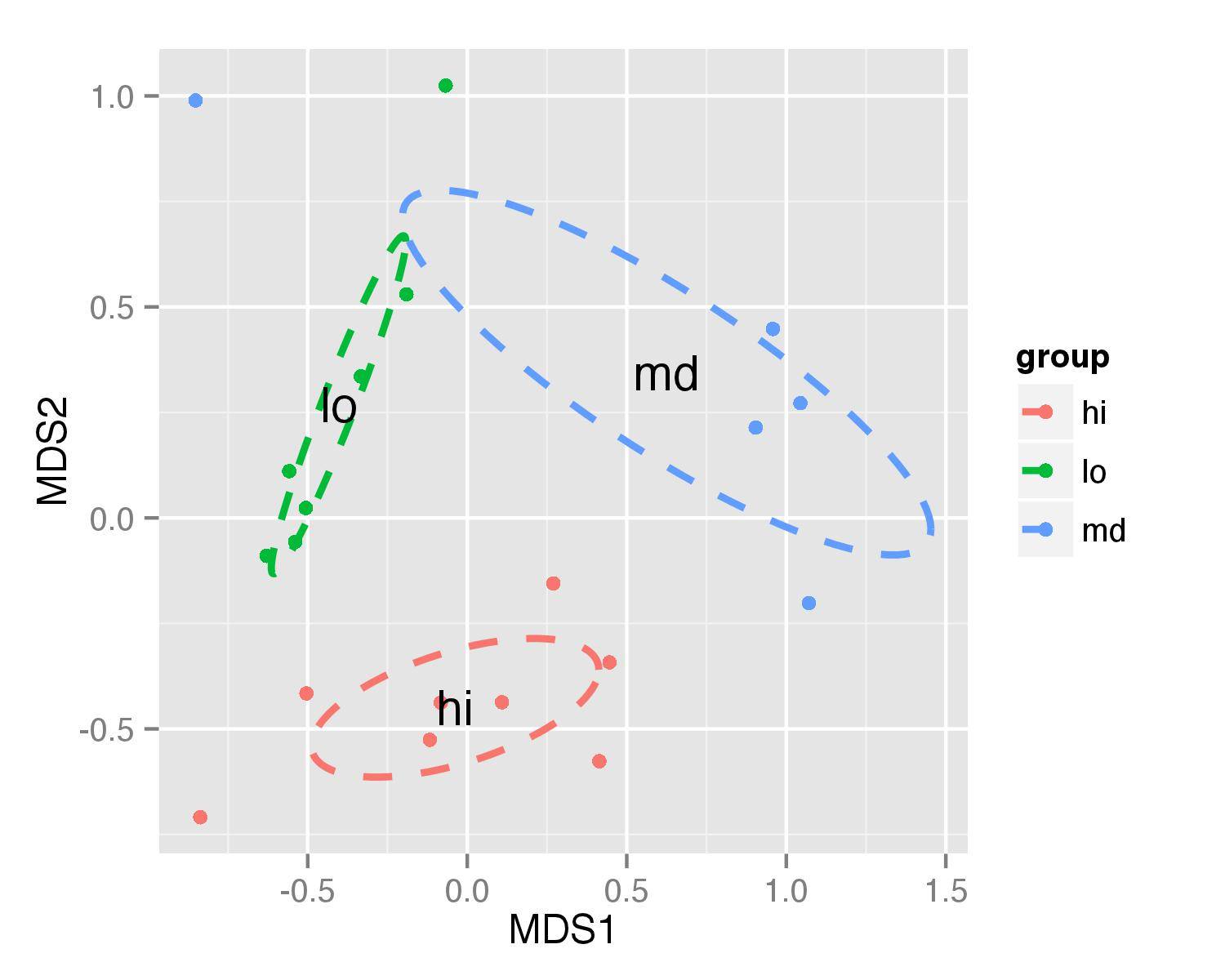
UPDATE-решение, которое работает в обоих случаях
во-первых, сделайте рамку данных NMDS с столбцом группы.
NMDS = data.frame(MDS1 = sol$points[,1], MDS2 = sol$points[,2],group=MyMeta$amt)
затем сохранить результат функции ordiellipse() как некоторые объект.
ord<-ordiellipse(sol, MyMeta$amt, display = "sites",
kind = "se", conf = 0.95, label = T)
фрейм данных df_ell содержит значения для отображения многоточий. Он вычисляется снова с помощью функции veganCovEllipse который скрыт в vegan пакета. Эта функция применяется к каждому уровню NMDS (group) и теперь использует аргументы, хранящиеся в это , это решение будет работать с различными параметрами, которые вы предоставляете для этой функции.
ggplot(data = NMDS, aes(MDS1, MDS2)) + geom_point(aes(color = group)) +
geom_path(data=df_ell, aes(x=NMDS1, y=NMDS2,colour=group), size=1, linetype=2)