построение результатов иерархической кластеризации на вершине матрицы данных в python
Как я могу построить дендрограмму прямо поверх матрицы значений, переупорядоченных соответствующим образом, чтобы отразить кластеризацию, в Python? Примером может служить следующий рисунок:
https://publishing-cdn.elifesciences.org/07103/elife-07103-fig6-figsupp1-v2.jpg
Я использую scipy.группа.дендрограмма, чтобы сделать мою дендрограмму и выполнить иерархическую кластеризацию на матрице данных. Как я могу отобразить данные в виде матрицы, где строки были упорядочены в отразите кластеризацию, вызванную разрезанием дендрограммы на определенном пороге, и построили ли дендрограмму рядом с матрицей? Я знаю, как построить дендрограмму в scipy, но не знаю, как построить матрицу интенсивности данных с правой шкалой рядом с ней.
любая помощь будет принята с благодарностью.
2 ответов
вопрос не определяет матрица очень хорошо: "матрица значений", "матрица данных". Я предполагаю, что вы имеете в виду расстояние матрицы. Другими словами, элемент D_ij в симметричном неотрицательном N-by-N расстояние матрицы D обозначает расстояние между двумя векторами объектов, x_i и x_j. Это верно?
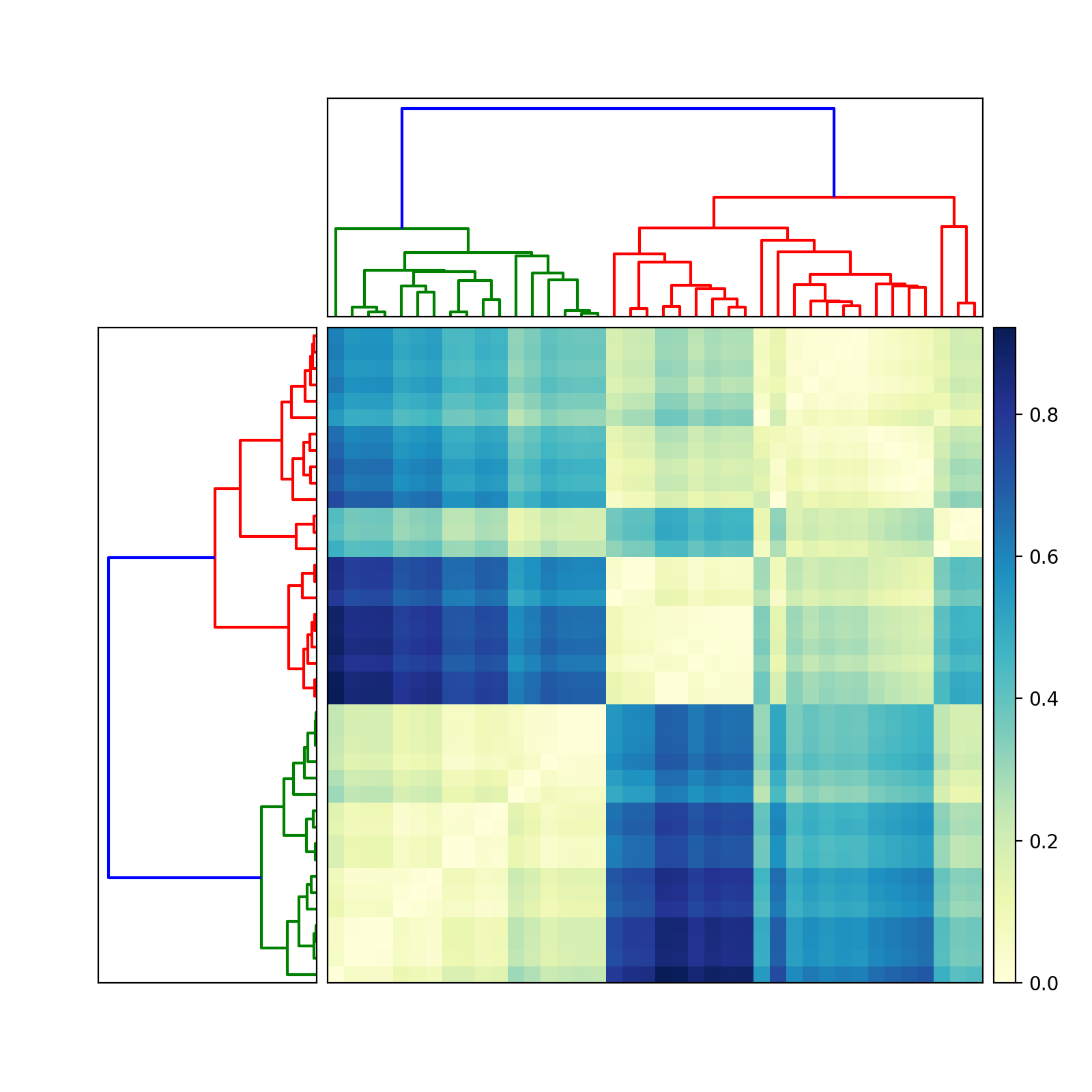
если да, то попробуйте это (отредактировано 13 июня 2010 года, чтобы отразить два разных дендрограммы):
import scipy
import pylab
import scipy.cluster.hierarchy as sch
from scipy.spatial.distance import squareform
# Generate random features and distance matrix.
x = scipy.rand(40)
D = scipy.zeros([40,40])
for i in range(40):
for j in range(40):
D[i,j] = abs(x[i] - x[j])
condensedD = squareform(D)
# Compute and plot first dendrogram.
fig = pylab.figure(figsize=(8,8))
ax1 = fig.add_axes([0.09,0.1,0.2,0.6])
Y = sch.linkage(condensedD, method='centroid')
Z1 = sch.dendrogram(Y, orientation='left')
ax1.set_xticks([])
ax1.set_yticks([])
# Compute and plot second dendrogram.
ax2 = fig.add_axes([0.3,0.71,0.6,0.2])
Y = sch.linkage(condensedD, method='single')
Z2 = sch.dendrogram(Y)
ax2.set_xticks([])
ax2.set_yticks([])
# Plot distance matrix.
axmatrix = fig.add_axes([0.3,0.1,0.6,0.6])
idx1 = Z1['leaves']
idx2 = Z2['leaves']
D = D[idx1,:]
D = D[:,idx2]
im = axmatrix.matshow(D, aspect='auto', origin='lower', cmap=pylab.cm.YlGnBu)
axmatrix.set_xticks([])
axmatrix.set_yticks([])
# Plot colorbar.
axcolor = fig.add_axes([0.91,0.1,0.02,0.6])
pylab.colorbar(im, cax=axcolor)
fig.show()
fig.savefig('dendrogram.png')
удачи! Дай мне знать, если тебе понадобится помощь.
Edit: для разных цветов настройте
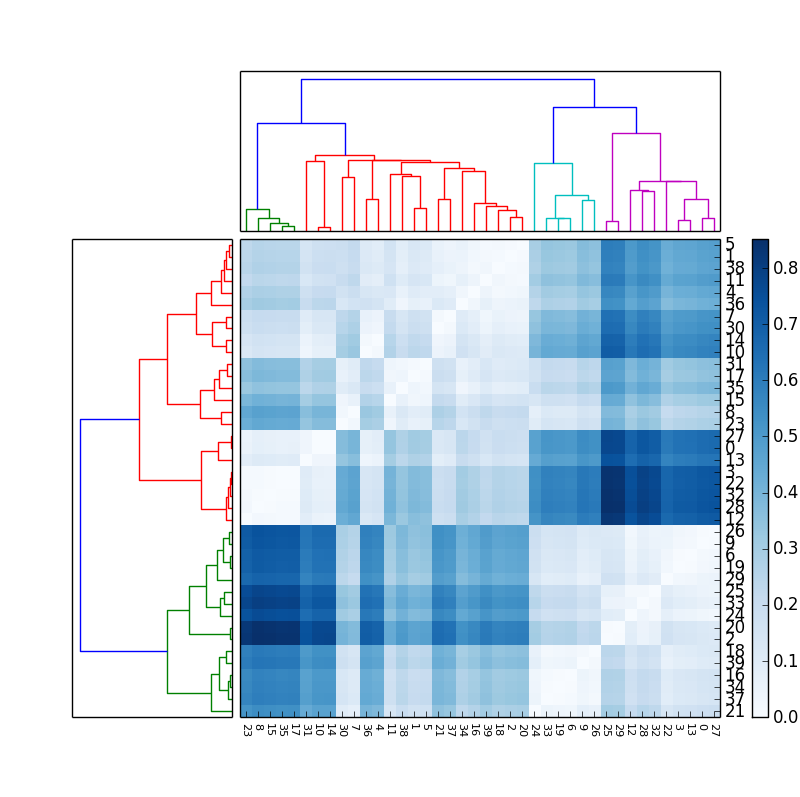
Если в дополнение к Матрице и дендрограмме требуется показать метки элементов, можно использовать следующий код, который показывает все метки, вращающие метки x и изменяющие размер шрифта, чтобы избежать перекрытия по оси X. Это требует перемещения цветовой панели, чтобы иметь пространство для y-меток:
axmatrix.set_xticks(range(40))
axmatrix.set_xticklabels(idx1, minor=False)
axmatrix.xaxis.set_label_position('bottom')
axmatrix.xaxis.tick_bottom()
pylab.xticks(rotation=-90, fontsize=8)
axmatrix.set_yticks(range(40))
axmatrix.set_yticklabels(idx2, minor=False)
axmatrix.yaxis.set_label_position('right')
axmatrix.yaxis.tick_right()
axcolor = fig.add_axes([0.94,0.1,0.02,0.6])
полученный результат таков (с другой цветовой картой):