При каких параметрах SVC и LinearSVC в scikit-learn эквивалентны?
прочитал этой теме о разнице между SVC() и LinearSVC() в scikit-учиться.
теперь у меня есть набор данных проблемы двоичной классификации(для такой проблемы разница стратегии Один к одному/один к отдыху между обеими функциями может быть проигнорирована.)
Я хочу попробовать, при каких параметрах эти 2 функции дадут мне тот же результат. Прежде всего, конечно, мы должны поставить kernel='linear' на SVC()
Однако я просто не мог получить то же самое результат от обеих функций. Я не смог найти ответ из документов, может ли кто-нибудь помочь мне найти эквивалентный набор параметров, который я ищу?
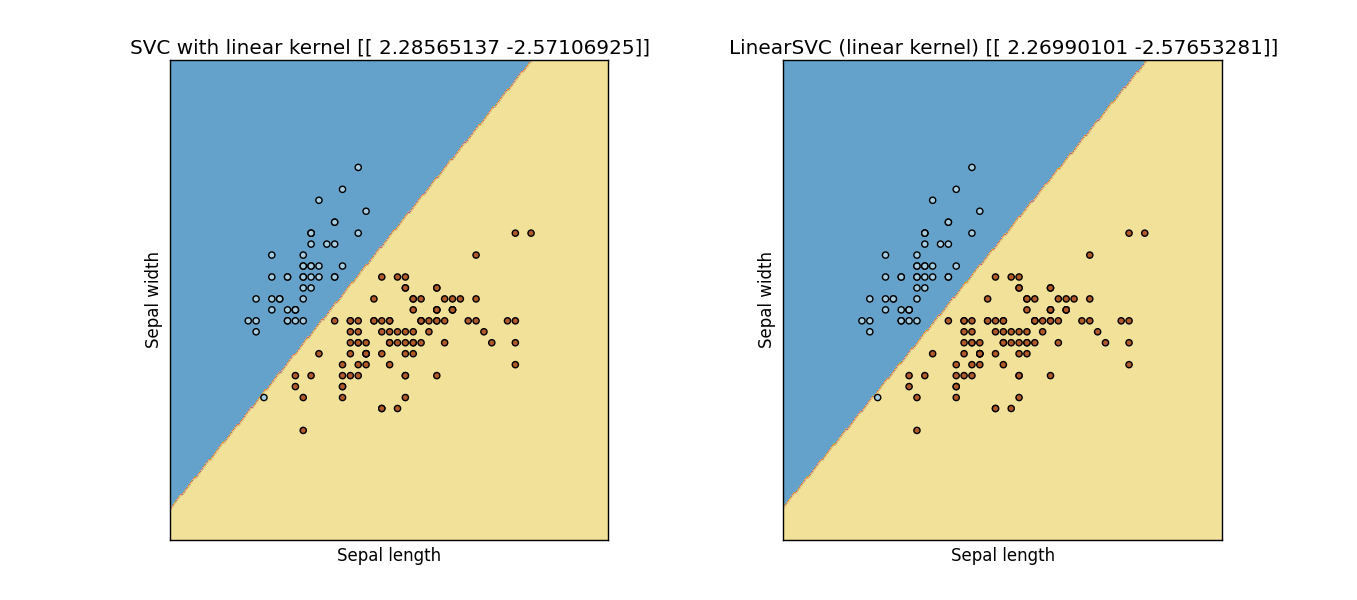
обновление: Я изменил следующий код из примера веб-сайта scikit-learn, и, по-видимому, они не совпадают:
import numpy as np
import matplotlib.pyplot as plt
from sklearn import svm, datasets
# import some data to play with
iris = datasets.load_iris()
X = iris.data[:, :2] # we only take the first two features. We could
# avoid this ugly slicing by using a two-dim dataset
y = iris.target
for i in range(len(y)):
if (y[i]==2):
y[i] = 1
h = .02 # step size in the mesh
# we create an instance of SVM and fit out data. We do not scale our
# data since we want to plot the support vectors
C = 1.0 # SVM regularization parameter
svc = svm.SVC(kernel='linear', C=C).fit(X, y)
lin_svc = svm.LinearSVC(C=C, dual = True, loss = 'hinge').fit(X, y)
# create a mesh to plot in
x_min, x_max = X[:, 0].min() - 1, X[:, 0].max() + 1
y_min, y_max = X[:, 1].min() - 1, X[:, 1].max() + 1
xx, yy = np.meshgrid(np.arange(x_min, x_max, h),
np.arange(y_min, y_max, h))
# title for the plots
titles = ['SVC with linear kernel',
'LinearSVC (linear kernel)']
for i, clf in enumerate((svc, lin_svc)):
# Plot the decision boundary. For that, we will assign a color to each
# point in the mesh [x_min, m_max]x[y_min, y_max].
plt.subplot(1, 2, i + 1)
plt.subplots_adjust(wspace=0.4, hspace=0.4)
Z = clf.predict(np.c_[xx.ravel(), yy.ravel()])
# Put the result into a color plot
Z = Z.reshape(xx.shape)
plt.contourf(xx, yy, Z, cmap=plt.cm.Paired, alpha=0.8)
# Plot also the training points
plt.scatter(X[:, 0], X[:, 1], c=y, cmap=plt.cm.Paired)
plt.xlabel('Sepal length')
plt.ylabel('Sepal width')
plt.xlim(xx.min(), xx.max())
plt.ylim(yy.min(), yy.max())
plt.xticks(())
plt.yticks(())
plt.title(titles[i])
plt.show()
результат: выход Рис. из предыдущего кода
1 ответов
в математическом смысле вам нужно установить:
SVC(kernel='linear', **kwargs) # by default it uses RBF kernel
и
LinearSVC(loss='hinge', **kwargs) # by default it uses squared hinge loss
другой элемент, который не может быть легко исправлен, увеличивается intercept_scaling на LinearSVC, поскольку в этой реализации смещение регуляризовано (что неверно в SVC и не должно быть истинным в SVM-таким образом это не SVM) - следовательно, они будут никогда быть точно равными (если смещение=0 для вашей проблемы), поскольку они предполагают две разные модели
- SVC :
1/2||w||^2 + C SUM xi_i - LinearSVC:
1/2||[w b]||^2 + C SUM xi_i
лично я считаю LinearSVC одной из ошибок sklearn developesr - этот класс просто не линейный SVM.
после увеличения масштабирования перехвата (до 10.0)
однако, если вы масштабируете его слишком много - он также потерпит неудачу, так как теперь допуск и количество итераций имеют решающее значение.
подведем итоги: LinearSVC не является линейным SVM, не используйте его, если не нужно.