Удалите строки со всеми или некоторыми NAs (отсутствующими значениями) в данных.рамка
Я хотел бы удалить строки в этом фрейме данных, которые:
a)содержат NAпо всем столбцам. Ниже приведен мой пример фрейма данных.
gene hsap mmul mmus rnor cfam
1 ENSG00000208234 0 NA NA NA NA
2 ENSG00000199674 0 2 2 2 2
3 ENSG00000221622 0 NA NA NA NA
4 ENSG00000207604 0 NA NA 1 2
5 ENSG00000207431 0 NA NA NA NA
6 ENSG00000221312 0 1 2 3 2
в принципе, я хотел бы получить фрейм данных, такой как следующий.
gene hsap mmul mmus rnor cfam
2 ENSG00000199674 0 2 2 2 2
6 ENSG00000221312 0 1 2 3 2
b)содержат NAs только в некоторых столбцах, поэтому я также могу получить этот результат:
gene hsap mmul mmus rnor cfam
2 ENSG00000199674 0 2 2 2 2
4 ENSG00000207604 0 NA NA 1 2
6 ENSG00000221312 0 1 2 3 2
15 ответов
также проверить complete.cases :
> final[complete.cases(final), ]
gene hsap mmul mmus rnor cfam
2 ENSG00000199674 0 2 2 2 2
6 ENSG00000221312 0 1 2 3 2
na.omit лучше просто удалить все NA'ы. complete.cases позволяет частичный выбор, включая только определенные столбцы фрейма данных:
> final[complete.cases(final[ , 5:6]),]
gene hsap mmul mmus rnor cfam
2 ENSG00000199674 0 2 2 2 2
4 ENSG00000207604 0 NA NA 1 2
6 ENSG00000221312 0 1 2 3 2
ваше решение не может работать. Если вы настаиваете на использовании is.na, то вы должны сделать что-то вроде:
> final[rowSums(is.na(final[ , 5:6])) == 0, ]
gene hsap mmul mmus rnor cfam
2 ENSG00000199674 0 2 2 2 2
4 ENSG00000207604 0 NA NA 1 2
6 ENSG00000221312 0 1 2 3 2
, но через complete.cases довольно намного яснее и быстрее.
попробовать na.omit(your.data.frame). Что касается второго вопроса, попробуйте разместить его как другой вопрос (для ясности).
Я предпочитаю следующий способ проверить, содержат ли строки NAs:
row.has.na <- apply(final, 1, function(x){any(is.na(x))})
Это возвращает логический вектор со значениями, обозначающими, есть ли NA в строке. Вы можете использовать его, чтобы увидеть, сколько строк вам придется сбрасывать:
sum(row.has.na)
и в конце концов бросают их
final.filtered <- final[!row.has.na,]
для фильтрации строк с определенной частью NAs это становится немного сложнее (например, вы можете подать " final[,5:6]", чтобы "применить"). Как правило, решение Joris Meys кажется более элегантный.
если вы любите труб (%>%),tidyrновое drop_na - ваш друг:
library(tidyr)
df %>% drop_na()
# gene hsap mmul mmus rnor cfam
# 2 ENSG00000199674 0 2 2 2 2
# 6 ENSG00000221312 0 1 2 3 2
df %>% drop_na(rnor, cfam)
# gene hsap mmul mmus rnor cfam
# 2 ENSG00000199674 0 2 2 2 2
# 4 ENSG00000207604 0 NA NA 1 2
# 6 ENSG00000221312 0 1 2 3 2
другой вариант, если вы хотите получить больший контроль над тем, как строки считаются недействительными, -
final <- final[!(is.na(final$rnor)) | !(is.na(rawdata$cfam)),]
используя выше, это:
gene hsap mmul mmus rnor cfam
1 ENSG00000208234 0 NA NA NA 2
2 ENSG00000199674 0 2 2 2 2
3 ENSG00000221622 0 NA NA 2 NA
4 ENSG00000207604 0 NA NA 1 2
5 ENSG00000207431 0 NA NA NA NA
6 ENSG00000221312 0 1 2 3 2
будет:
gene hsap mmul mmus rnor cfam
1 ENSG00000208234 0 NA NA NA 2
2 ENSG00000199674 0 2 2 2 2
3 ENSG00000221622 0 NA NA 2 NA
4 ENSG00000207604 0 NA NA 1 2
6 ENSG00000221312 0 1 2 3 2
...где удаляется только строка 5, так как это единственная строка, содержащая NAs для обоих rnor и cfam. Логика boolean может быть изменена в соответствии с определенными требованиями.
Если вы хотите контролировать, сколько NAs допустимы для каждой строки, попробуйте эту функцию. Для многих наборов данных опроса слишком много пустых ответов на вопросы могут испортить результаты. Таким образом, они удаляются после определенного порога. Эта функция позволит вам выбрать, сколько NAs может иметь строка, прежде чем она будет удалена:
delete.na <- function(DF, n=0) {
DF[rowSums(is.na(DF)) <= n,]
}
по умолчанию он устранит все NAs:
delete.na(final)
gene hsap mmul mmus rnor cfam
2 ENSG00000199674 0 2 2 2 2
6 ENSG00000221312 0 1 2 3 2
или укажите максимальное разрешенное количество NAs:
delete.na(final, 2)
gene hsap mmul mmus rnor cfam
2 ENSG00000199674 0 2 2 2 2
4 ENSG00000207604 0 NA NA 1 2
6 ENSG00000221312 0 1 2 3 2
это вернет строки, которые имеют по крайней мере одно значение, отличное от NA.
final[rowSums(is.na(final))<length(final),]
это вернет строки, которые имеют по крайней мере два значения, отличных от NA.
final[rowSums(is.na(final))<(length(final)-1),]
используя пакет dplyr, мы можем фильтровать NA следующим образом:
dplyr::filter(df, !is.na(columnname))
мы также можем использовать функцию подмножества для этого.
finalData<-subset(data,!(is.na(data["mmul"]) | is.na(data["rnor"])))
Это даст только те строки, которые не имеют NA как в mmul, так и в rnor
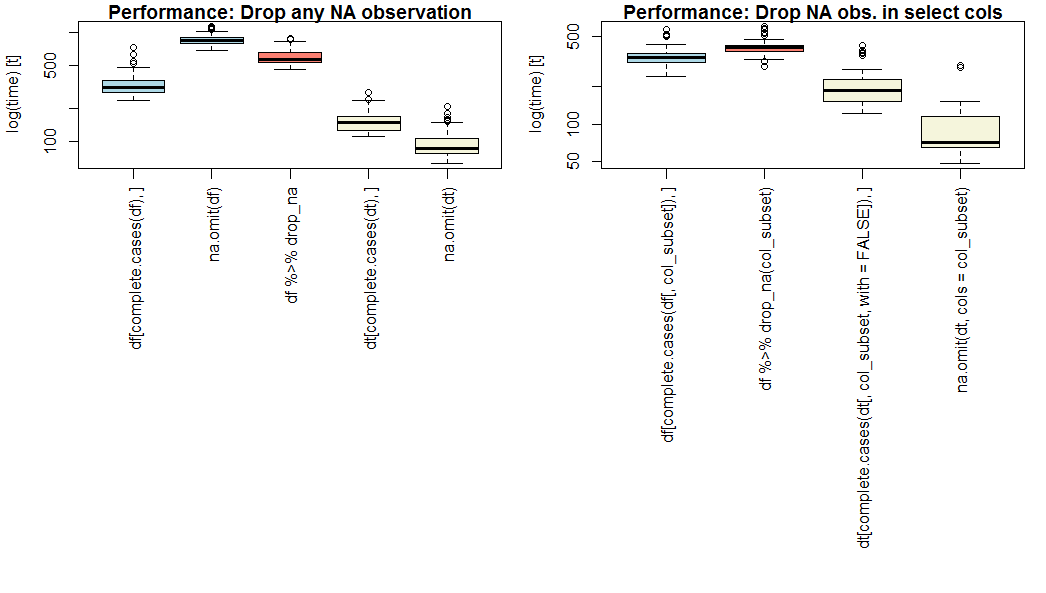
если производительность является приоритетом, используйте data.table и na.omit() с дополнительным парам cols=.
na.omit.data.table является самым быстрым на моем бенчмарке (см. ниже), будь то для всех столбцов или для выбранных столбцов (OP question part 2).
если вы не хотите использовать data.table используйте complete.cases().
на ваниль data.frame, complete.cases быстрее na.omit() или dplyr::drop_na(). Заметьте, что na.omit.data.frame не поддержка cols=.
результат теста
вот сравнение базы (синий), dplyr (розовый), и data.table (желтый) методы для удаления всех или выбора отсутствующих наблюдений, на условном наборе данных из 1 миллиона наблюдений 20 числовых переменных с независимой вероятностью 5% отсутствия и подмножеством из 4 переменных для части 2.
ваши результаты могут варьироваться в зависимости от длины, ширины и разреженности вашего конкретного набор данных.
Примечание логарифмической шкале по оси Y.
тест-скрипт
#------- Adjust these assumptions for your own use case ------------
row_size <- 1e6L
col_size <- 20 # not including ID column
p_missing <- 0.05 # likelihood of missing observation (except ID col)
col_subset <- 18:21 # second part of question: filter on select columns
#------- System info for benchmark ----------------------------------
R.version # R version 3.4.3 (2017-11-30), platform = x86_64-w64-mingw32
library(data.table); packageVersion('data.table') # 1.10.4.3
library(dplyr); packageVersion('dplyr') # 0.7.4
library(tidyr); packageVersion('tidyr') # 0.8.0
library(microbenchmark)
#------- Example dataset using above assumptions --------------------
fakeData <- function(m, n, p){
set.seed(123)
m <- matrix(runif(m*n), nrow=m, ncol=n)
m[m<p] <- NA
return(m)
}
df <- cbind( data.frame(id = paste0('ID',seq(row_size)),
stringsAsFactors = FALSE),
data.frame(fakeData(row_size, col_size, p_missing) )
)
dt <- data.table(df)
par(las=3, mfcol=c(1,2), mar=c(22,4,1,1)+0.1)
boxplot(
microbenchmark(
df[complete.cases(df), ],
na.omit(df),
df %>% drop_na,
dt[complete.cases(dt), ],
na.omit(dt)
), xlab='',
main = 'Performance: Drop any NA observation',
col=c(rep('lightblue',2),'salmon',rep('beige',2))
)
boxplot(
microbenchmark(
df[complete.cases(df[,col_subset]), ],
#na.omit(df), # col subset not supported in na.omit.data.frame
df %>% drop_na(col_subset),
dt[complete.cases(dt[,col_subset,with=FALSE]), ],
na.omit(dt, cols=col_subset) # see ?na.omit.data.table
), xlab='',
main = 'Performance: Drop NA obs. in select cols',
col=c('lightblue','salmon',rep('beige',2))
)
для вашего первого вопроса у меня есть код, с которым мне удобно избавиться от всех NAs. Спасибо за @Gregor, чтобы сделать его проще.
final[!(rowSums(is.na(final))),]
для второго вопроса код является просто чередованием с предыдущим решением.
final[as.logical((rowSums(is.na(final))-5)),]
обратите внимание, что -5-это количество столбцов в ваших данных. Это устранит строки со всеми NAs, так как суммы строк складываются до 5, и они становятся нулями после вычитания. На этот раз ас.логика необходима.
Я синтезатор:). Здесь я объединил ответы в одну функцию:
#' keep rows that have a certain number (range) of NAs anywhere/somewhere and delete others
#' @param df a data frame
#' @param col restrict to the columns where you would like to search for NA; eg, 3, c(3), 2:5, "place", c("place","age")
#' \cr default is NULL, search for all columns
#' @param n integer or vector, 0, c(3,5), number/range of NAs allowed.
#' \cr If a number, the exact number of NAs kept
#' \cr Range includes both ends 3<=n<=5
#' \cr Range could be -Inf, Inf
#' @return returns a new df with rows that have NA(s) removed
#' @export
ez.na.keep = function(df, col=NULL, n=0){
if (!is.null(col)) {
# R converts a single row/col to a vector if the parameter col has only one col
# see https://radfordneal.wordpress.com/2008/08/20/design-flaws-in-r-2-%E2%80%94-dropped-dimensions/#comments
df.temp = df[,col,drop=FALSE]
} else {
df.temp = df
}
if (length(n)==1){
if (n==0) {
# simply call complete.cases which might be faster
result = df[complete.cases(df.temp),]
} else {
# credit: http://stackoverflow.com/a/30461945/2292993
log <- apply(df.temp, 2, is.na)
logindex <- apply(log, 1, function(x) sum(x) == n)
result = df[logindex, ]
}
}
if (length(n)==2){
min = n[1]; max = n[2]
log <- apply(df.temp, 2, is.na)
logindex <- apply(log, 1, function(x) {sum(x) >= min && sum(x) <= max})
result = df[logindex, ]
}
return(result)
}
предполагая, что dat в качестве фрейма данных ожидаемый результат может быть достигнут с помощью
1.rowSums
> dat[!rowSums((is.na(dat))),]
gene hsap mmul mmus rnor cfam
2 ENSG00000199674 0 2 2 2 2
6 ENSG00000221312 0 1 2 3 2
2.lapply
> dat[!Reduce('|',lapply(dat,is.na)),]
gene hsap mmul mmus rnor cfam
2 ENSG00000199674 0 2 2 2 2
6 ENSG00000221312 0 1 2 3 2
delete.dirt <- function(DF, dart=c('NA')) {
dirty_rows <- apply(DF, 1, function(r) !any(r %in% dart))
DF <- DF[dirty_rows, ]
}
mydata <- delete.dirt(mydata)
выше функция удаляет все строки из фрейма данных, который имеет " NA " в любом столбце и возвращает результирующие данные. Если вы хотите проверить несколько значений типа NA и ? изменить dart=c('NA') в функции param to dart=c('NA', '?')
Я предполагаю, что это может быть более элегантно решена таким образом
m <- matrix(1:25, ncol = 5)
m[c(1, 6, 13, 25)] <- NA
df <- data.frame(m)
library(dplyr)
df %>%
filter_all(any_vars(is.na(.)))
#> X1 X2 X3 X4 X5
#> 1 NA NA 11 16 21
#> 2 3 8 NA 18 23
#> 3 5 10 15 20 NA