Улучшение компоновки графика Python NetworkX
У меня возникли проблемы с визуализацией графиков, созданных с помощью python-networkx, я хочу уменьшить беспорядок и регулировать расстояние между узлами (я также пробовал spring_layout, он просто раскладывает узлы эллиптическим способом). Посоветуйте, пожалуйста.
код:
nx.draw_networkx_edges(G, pos, edgelist=predges, edge_color='red', arrows=True)
nx.draw_networkx_edges(G, pos, edgelist=black_edges, arrows=False, style='dashed')
# label fonts
nx.draw_networkx_labels(G,pos,font_size=7,font_family='sans-serif')
nx.draw_networkx_edge_labels(G,pos,q_list,label_pos=0.3)
4 ответов
в networkx стоит проверить алгоритмы рисования графов, предоставляемые graphviz via nx.graphviz_layout.
у меня был хороший успех с neato но другие возможные входные данные -
dot- "иерархические" или слоистые рисунки направленных графов. Это инструмент по умолчанию для использования, если ребра имеют направленность.neato- "весна" модели макеты. Это инструмент по умолчанию используйте, если график не слишком велик (около 100 узлов), и вы ничего не знаете об этом. Neato пытается минимизировать глобальную энергетическую функцию, эквивалентную статистическому многомерному масштабированию.fdp- макеты "пружинной модели" похожи на макеты neato, но делают это, уменьшая силы, а не работая с энергией.sfdp- многомасштабная версия fdp для компоновки больших диаграммы.twopi- радиальные макеты, после Грэма Уиллса 97. Узлы размещаются на концентрических окружностях в зависимости от их расстояния от заданного корневого узла.circo- круговой макет, после шести и Толлиса 99, Кауфмана и Визе 02. Это подходит для некоторых диаграмм нескольких циклических структур, таких как определенные телекоммуникационные сети.
в общем, графа - это трудно проблема. Если этих алгоритмов недостаточно, вам придется написать свои собственные или иметь части рисования networkx по отдельности.
у вас много данных на графике, поэтому будет трудно удалить беспорядок.
Я предлагаю вам использовать любой стандартный формат. Вы сказали, что использовали spring_layout. Я предлагаю вам попробовать еще раз, но на этот раз с помощью weight атрибут при добавлении ребер.
например:
import networkx as nx
G = nx.Graph();
G.add_node('A')
G.add_node('B')
G.add_node('C')
G.add_node('D')
G.add_edge('A','B',weight=1)
G.add_edge('C','B',weight=1)
G.add_edge('B','D',weight=30)
pos = nx.spring_layout(G,scale=2)
nx.draw(G,pos,font_size=8)
plt.show()
кроме того, вы можете использовать параметр scale для увеличения глобального расстояния между узлами.
чтобы ответить на ваш вопрос, как регулировать расстояние между узлами, мы его расширим зацепило это:
если вы рисуете график через бэкэнд Graphviz и когда вы затем используете fdp алгоритм, вы можете настроить расстояние между узлами по edge атрибут len.
вот пример кода, как нарисовать график G и сохранить в файле Graphviz gvfile С более широким расстоянием между узлами (по умолчанию расстояние для fdp is 0.3):
A = nx.to_agraph(G)
A.edge_attr.update(len=3)
A.write(gv_file_name)
два комментария:
- обычно рекомендуется настроить
lenС количеством узлов в графе. - на
lenатрибут распознается толькоfdpи , а не, например,.
Я нашел это полезным для быстрой визуализации данных взаимодействия, полученных в виде CSV-файла из PostgreSQL. [Вывод ниже переформатирован для удобства чтения.]
## PSQL ['DUMMY' DATA]:
[interactions_practice]# \copy (SELECT gene_1, gene_2 FROM interactions
WHERE gene_1 in (SELECT gene_2 FROM interactions))
TO '/tmp/a.csv' WITH CSV -- << note: no terminating ";" for this query
## BASH:
[victoria@victoria ~]$ cat /tmp/a.csv
APC,TP73
BARD1,BRCA1
BARD1,ESR1
BARD1,KRAS2
BARD1,SLC22A18
BARD1,TP53
BRCA1,BRCA2
BRCA1,CHEK2
BRCA1,MLH1
BRCA1,PHB
BRCA2,CHEK2
BRCA2,TP53
CASP8,ESR1
CASP8,KRAS2
CASP8,PIK3CA
CASP8,SLC22A18
CDK2,CDKN1A
CHEK2,CDK2
ESR1,BRCA1
ESR1,KRAS2
ESR1,PPM1D
ESR1,SLC22A18
KRAS2,BRCA1
MLH1,CHEK2
MLH1,PMS2
PIK3CA,BRCA1
PIK3CA,ESR1
PIK3CA,RB1CC1
PIK3CA,SLC22A18
PMS2,TP53
PTEN,BRCA1
PTEN,MLH3
RAD51,BRCA1
RB1CC1,SLC22A18
SLC22A18,BRCA1
TP53,PTEN
## PYTHON 3.5 VENV (ANACONDA):
>>> import networkx as nx
>>> import pylab as plt
>>> G = nx.read_edgelist("/tmp/a.csv", delimiter=",")
>>> G.edges()
[('CDKN1A', 'CDK2'), ('MLH3', 'PTEN'), ('TP73', 'APC'), ('CHEK2', 'MLH1'),
('CHEK2', 'BRCA2'), ('CHEK2', 'CDK2'), ('CHEK2', 'BRCA1'), ('BRCA2', 'TP53'),
('BRCA2', 'BRCA1'), ('KRAS2', 'CASP8'), ('KRAS2', 'ESR1'), ('KRAS2', 'BRCA1'),
('KRAS2', 'BARD1'), ('PPM1D', 'ESR1'), ('BRCA1', 'PHB'), ('BRCA1', 'ESR1'),
('BRCA1', 'PIK3CA'), ('BRCA1', 'PTEN'), ('BRCA1', 'MLH1'), ('BRCA1', 'SLC22A18'),
('BRCA1', 'BARD1'), ('BRCA1', 'RAD51'), ('CASP8', 'ESR1'), ('CASP8', 'SLC22A18'),
('CASP8', 'PIK3CA'), ('TP53', 'PMS2'), ('TP53', 'PTEN'), ('TP53', 'BARD1'),
('PMS2', 'MLH1'), ('PIK3CA', 'SLC22A18'), ('PIK3CA', 'ESR1'), ('PIK3CA', 'RB1CC1'),
('SLC22A18', 'ESR1'), ('SLC22A18', 'RB1CC1'), ('SLC22A18', 'BARD1'), ('BARD1', 'ESR1')]
>>> G.number_of_edges()
36
>>> G.nodes()
['CDKN1A', 'MLH3', 'TP73', 'CHEK2', 'BRCA2', 'KRAS2', 'CDK2', 'PPM1D', 'BRCA1',
'CASP8', 'TP53', 'PMS2', 'RAD51', 'PIK3CA', 'MLH1', 'SLC22A18', 'BARD1', 'PHB', 'APC', 'ESR1', 'RB1CC1', 'PTEN']
>>> G.number_of_nodes()
22
>>> from networkx.drawing.nx_agraph import graphviz_layout
>>> ## nx.draw(G, pos=graphviz_layout(G))
## DUE TO AN UNIDENTIFIED BUG, I GET THIS ERROR THE FIRST TIME RUNNING THIS
## COMMAND; JUST RE-RUN IT:
>>> nx.draw(G, pos=graphviz_layout(G), node_size=1200, node_color='lightblue',
linewidths=0.25, font_size=10, font_weight='bold', with_labels=True)
QGtkStyle could not resolve GTK. Make sure you have installed the proper libraries.
>>> nx.draw(G, pos=graphviz_layout(G), node_size=1200, node_color='lightblue',
linewidths=0.25, font_size=10, font_weight='bold', with_labels=True)
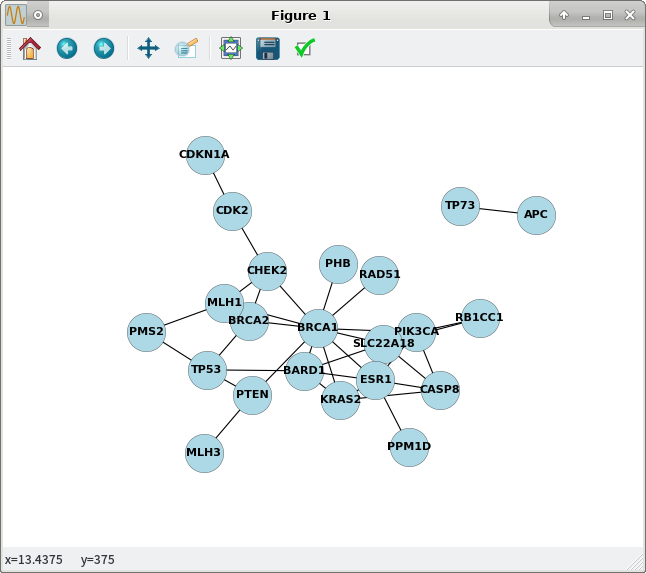
>>> plt.show() ## plot1.png [opens in matplotlib popup window] attached
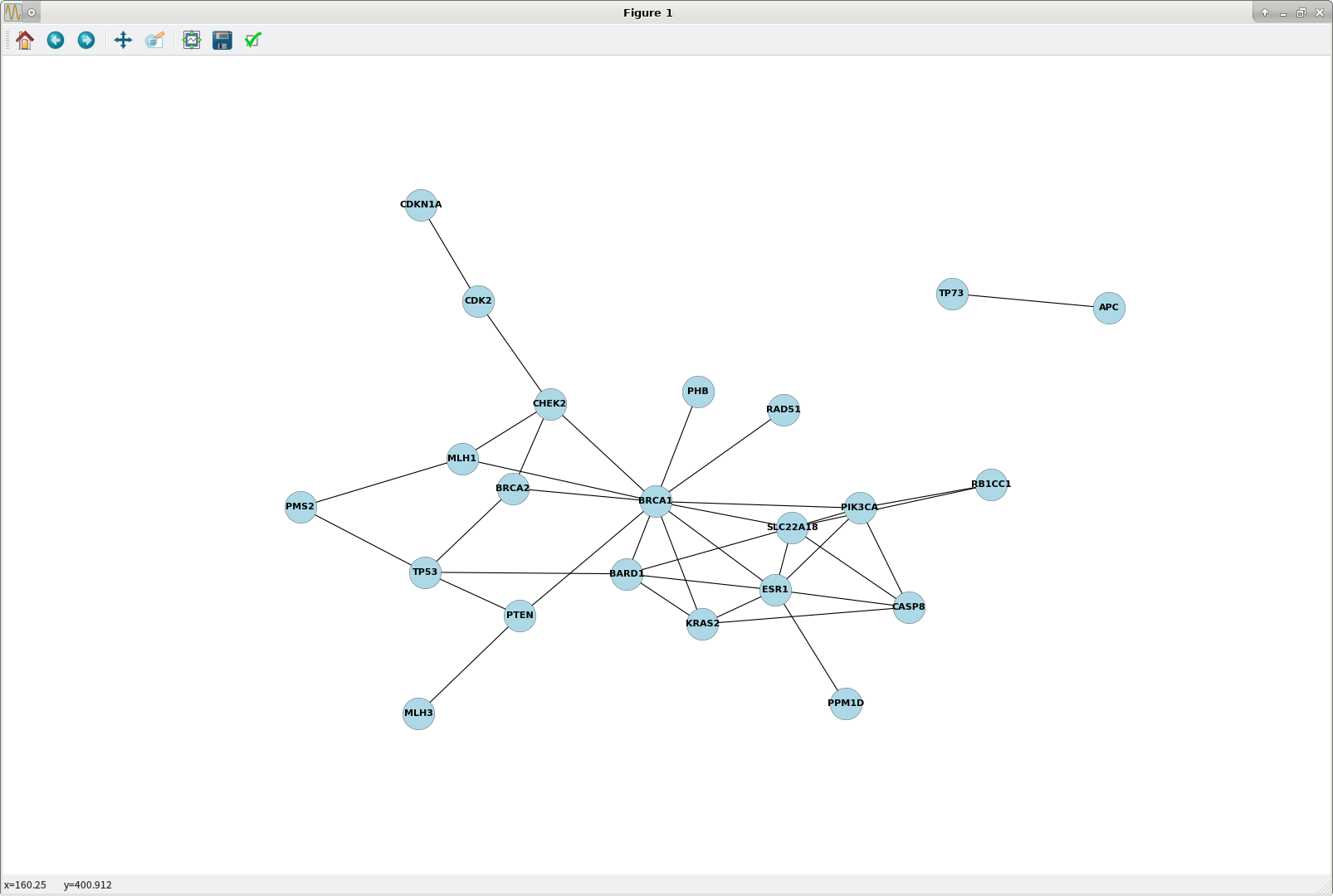
трудно уменьшить перегрузку в этих статических графиках networkx / matplotlib; один обходной путь-увеличить размер фигуры в этом StackOverflow Q / A: высокое разрешение изображения графика с помощью NetworkX и Matplotlib :
>>> plt.figure(figsize=(20,14))
<matplotlib.figure.Figure object at 0x7f1b65ea5e80>
>>> nx.draw(G, pos=graphviz_layout(G), node_size=1200, node_color='lightblue',
linewidths=0.25, font_size=10, font_weight='bold', with_labels=True, dpi=1000)
>>> plt.show() ## plot2.png attached
## RESET OUTPUT FIGURE SIZE TO SYSTEM DEFAULT:
>>> plt.figure()
<matplotlib.figure.Figure object at 0x7f1b454f1588>
бонус -- кратчайший путь:
>>> nx.dijkstra_path(G, 'CDKN1A', 'MLH3')
['CDKN1A', 'CDK2', 'CHEK2', 'BRCA1', 'PTEN', 'MLH3']