Как построить дендрограммы с большими наборами данных?
Я использую пакет ape (анализ филогенетики и эволюции) в R, который имеет функциональность рисования дендрограммы. Я использую следующие команды для чтения данных в формате Newick и рисования дендрограммы с помощью функции plot:
library("ape")
gcPhylo <-read.tree(file = "gc.tree")
plot(gcPhylo, show.node.label = TRUE)
поскольку набор данных довольно большой, невозможно увидеть какие-либо детали на нижних уровнях дерева. Я вижу только черные области, но никаких деталей. Я вижу лишь несколько уровней. сверху, а потом никаких подробностей.
мне было интересно, есть ли возможность масштабирования функции графика. Я попытался ограничить область с помощью xLim и yLim, однако они просто ограничивают область и не масштабируют, чтобы сделать детали видимыми. Либо масштабирование, либо отображение деталей без масштабирования решит мою проблему.
Я также признателен за знание любого другого пакета, функции или инструмента, которые помогут мне преодолеть проблему.
спасибо.
2 ответов
на cut функция, описанная в другом ответе, является очень хорошим решением; если вы хотите сохранить все дерево на одной странице для интерактивного исследования, вы также можете построить график на большой странице в формате PDF.
результирующий PDF векторизован, так что вы можете увеличить близко с вашим любимым PDF viewer без потери разрешения.
вот пример того, как направить вывод графика в PDF:
# Open a PDF for plotting; units are inches by default
pdf("/path/to/a/pdf/file.pdf", width=40, height=15)
# Do some plotting
plot(gcPhylo)
# Close the PDF file's associated graphics device (necessary to finalize the output)
dev.off()
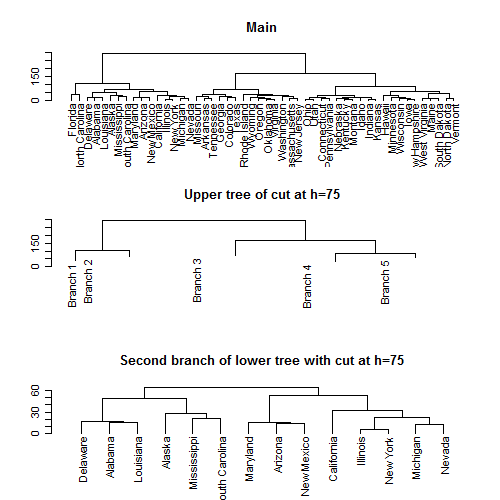
можно cut дендрограмма на заданной высоте и построение элементов:
сначала создайте кластеризацию с помощью встроенного набора данных USArrests. Затем преобразуйте в dendrogram:
hc <- hclust(dist(USArrests))
hcd <- as.dendrogram(hc)
далее используйте cut.dendrogram вырезать на заданной высоте, в этом случае h=75. Это создает список дендрограммы для upper бит распила, и список dendograms, по одному для каждой branch ниже:
par(mfrow=c(3,1))
plot(hcd, main="Main")
plot(cut(hcd, h=75)$upper,
main="Upper tree of cut at h=75")
plot(cut(hcd, h=75)$lower[[2]],
main="Second branch of lower tree with cut at h=75")