Как построить двудольный граф в R
Как построить сеть типа двудольные в R? Похожие на это:
У меня есть аналогичные данные, но с Весами как для генов, так и для болезней и торс. Эта сеть является примером. У меня разные качества. Я последовал за а ссылке здесь. Но из-за моих небольших знаний в этой теме, я не мог получить много из него. Заранее спасибо за любую помощь.
3 ответов
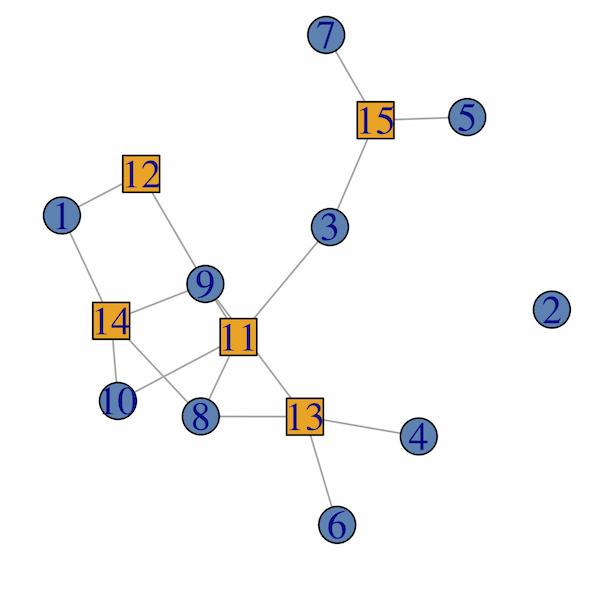
С ?bipartite_graph справка:
двудольные графы имеют атрибут вершины типа в igraph, это булево и ложно для вершин первого рода и истинно для вершин второго рода.
так что вы могли бы сделать что-то подобное (igraph 1.0.1):
library(igraph)
set.seed(123)
# generate random bipartite graph.
g <- sample_bipartite(10, 5, p=.4)
# check the type attribute:
V(g)$type
# define color and shape mappings.
col <- c("steelblue", "orange")
shape <- c("circle", "square")
plot(g,
vertex.color = col[as.numeric(V(g)$type)+1],
vertex.shape = shape[as.numeric(V(g)$type)+1]
)
проверить тоже ?bipartite.
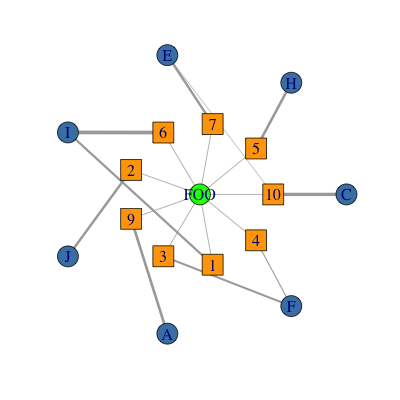
используя пример, предоставленный OP в комментариях. Поскольку граф является многопартийным и заданным в предоставленном формате данных я бы сначала создал двудольный граф, а затем добавил дополнительные ребра. Обратите внимание, что хотя результирующий график возвращает TRUE для is_bipartite() аргумент типа указан как числовой вместо логического и может неправильно работать с другими двудольными функциями.
set.seed(123)
V1 <- sample(LETTERS[1:10], size = 10, replace = TRUE)
V2 <- sample(1:10, size = 10, replace = TRUE)
d <- data.frame(V1 = V1, V2 = V2, weights = runif(10))
d
> d
V1 V2 weights
1 C 10 0.8895393
2 H 5 0.6928034
3 E 7 0.6405068
4 I 6 0.9942698
5 J 2 0.6557058
6 A 9 0.7085305
7 F 3 0.5440660
8 I 1 0.5941420
9 F 4 0.2891597
10 E 10 0.1471136
g <- graph_from_data_frame(d, directed = FALSE)
V(g)$label <- V(g)$name # set labels.
# create a graph connecting central node FOO to each V2.
e <- expand.grid(V2 = unique(d$V2), V2 = "FOO")
> e
V2 V2
1 10 FOO
2 5 FOO
3 7 FOO
4 6 FOO
5 2 FOO
6 9 FOO
7 3 FOO
8 1 FOO
9 4 FOO
g2 <- graph.data.frame(e, directed = FALSE)
# join the two graphs.
g <- g + g2
# set type.
V(g)$type <- 1
V(g)[name %in% 1:10]$type <- 2
V(g)[name %in% "FOO"]$type <- 3
V(g)$type
> V(g)$type
[1] 1 1 1 1 1 1 1 2 2 2 2 2 2 2 2 2 3
col <- c("steelblue", "orange", "green")
shape <- c("circle", "square", "circle")
library(rTRM) # Bioconductor package containing layout.concentric()
# the fist element in the list for concentric is the central node.
l <- layout.concentric(g, concentric = list("FOO", 1:10, LETTERS[1:10]))
plot(g,
layout = l,
vertex.color = col[V(g)$type],
vertex.shape = shape[V(g)$type],
edge.width = E(g)$weights * 5 # optional, plot edges width proportional to weights.
)
функции layout.concentric() в (мой) пакет rTRM, от программе bioconductor. Это действительно простая реализация, которую я написал, чтобы сделать именно то, что ты хочешь. Я не совсем уверен, что последний igraph версия имеет ту же функциональность, хотя (может быть).
для примера, который вы предоставили, я бы рекомендовал использовать x и y атрибуты для визуализации двудольного графа. Например:
V(g)$x <- c(1, 1, 1, 2, 2, 2, 2)
V(g)$y <- c(3, 2, 1, 3.5, 2.5, 1.5, 0.5)
V(g)$shape <- shape[as.numeric(V(g)$type) + 1]
V(g)$color <- c('red', 'blue', 'green', 'steelblue', 'steelblue', 'steelblue', 'steelblue')
E(g)$color <- 'gray'
E(g)$color[E(g)['A' %--% V(g)]] <- 'red'
E(g)$color[E(g)['B' %--% V(g)]] <- 'blue'
E(g)$color[E(g)['C' %--% V(g)]] <- 'green'
plot(g)
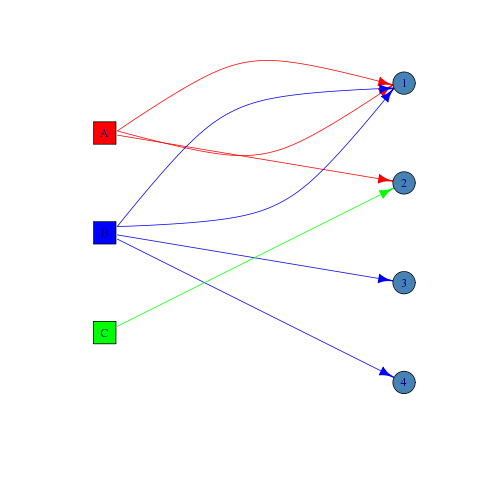
EDIT: добавлен код для придания вершинам и ребрам разных цветов для ясности.
или вы можете использовать multigraph пакета.
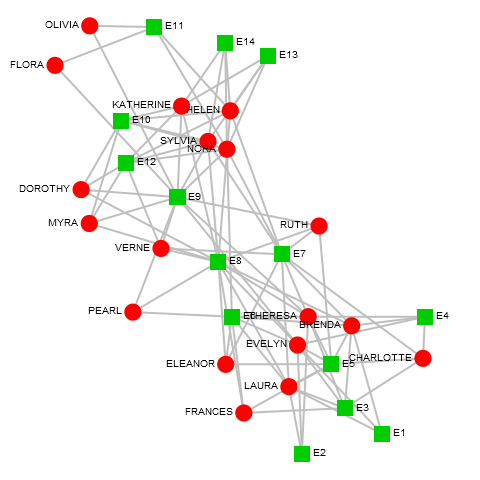
swomen <- read.dl(file = "http://moreno.ss.uci.edu/davis.dat")
bmgraph(swomen, layout = "force", seed = 1, cex = 3, tcex = .8, pch = c(19, 15), lwd = 2,
+ vcol = 2:3, ecol = 8, rot = 65)
это может произвести биномиальную проекцию двухрежимного набора данных