Как решить проблемы с памятью при многопроцессорной обработке с помощью пула.map()?
я написал программу (ниже):
- читать большой текстовый файл как
pandas dataframe - затем
groupbyиспользование определенного значения столбца для разделения данных и хранения в виде списка кадров данных. - затем передайте данные в
multiprocess Pool.map()для обработки каждого фрейма данных параллельно.
все в порядке, программа хорошо работает на моем небольшом тестовом наборе данных. Но, когда я передаю свои большие данные (около 14 ГБ), потребление памяти экспоненциально увеличивает, а затем замораживает компьютер или погибает (в кластере HPC).
я добавил коды для очистки памяти, как только данные/переменные не полезно. Я также закрываю бассейн, как только это будет сделано. Все еще с 14 ГБ ввода я ожидал только 2 * 14 ГБ нагрузки на память, но похоже, что много происходит. Я также попытался настроить с помощью chunkSize and maxTaskPerChild, etc но я не вижу никакой разницы в оптимизации как в тесте, так и в большом файле.
Я думаю, что улучшения в этом коде is / требуются в этой позиции кода, когда я начинаю multiprocessing.
p = Pool(3) # number of pool to run at once; default at 1
result = p.map(matrix_to_vcf, list(gen_matrix_df_list.values()))
но я отправляю весь код.
тестовый пример: я создал тестовый файл ("genome_matrix_final-chr1234-1mb.txt") до 250 МБ и запустил программу. Когда я проверяю Системный монитор, я вижу, что потребление памяти увеличилось примерно на 6 ГБ. Я не совсем понимаю, почему так много места в памяти занимает файл 250 mb плюс некоторые выходы. Я поделился этим файлом через drop box, если это поможет вижу настоящую проблему. https://www.dropbox.com/sh/coihujii38t5prd/AABDXv8ACGIYczeMtzKBo0eea?dl=0
может кто-нибудь подсказать, как я могу избавиться от проблемы?
мой скрипт python:
#!/home/bin/python3
import pandas as pd
import collections
from multiprocessing import Pool
import io
import time
import resource
print()
print('Checking required modules')
print()
''' change this input file name and/or path as need be '''
genome_matrix_file = "genome_matrix_final-chr1n2-2mb.txt" # test file 01
genome_matrix_file = "genome_matrix_final-chr1234-1mb.txt" # test file 02
#genome_matrix_file = "genome_matrix_final.txt" # large file
def main():
with open("genome_matrix_header.txt") as header:
header = header.read().rstrip('n').split('t')
print()
time01 = time.time()
print('starting time: ', time01)
'''load the genome matrix file onto pandas as dataframe.
This makes is more easy for multiprocessing'''
gen_matrix_df = pd.read_csv(genome_matrix_file, sep='t', names=header)
# now, group the dataframe by chromosome/contig - so it can be multiprocessed
gen_matrix_df = gen_matrix_df.groupby('CHROM')
# store the splitted dataframes as list of key, values(pandas dataframe) pairs
# this list of dataframe will be used while multiprocessing
gen_matrix_df_list = collections.OrderedDict()
for chr_, data in gen_matrix_df:
gen_matrix_df_list[chr_] = data
# clear memory
del gen_matrix_df
'''Now, pipe each dataframe from the list using map.Pool() '''
p = Pool(3) # number of pool to run at once; default at 1
result = p.map(matrix_to_vcf, list(gen_matrix_df_list.values()))
del gen_matrix_df_list # clear memory
p.close()
p.join()
# concat the results from pool.map() and write it to a file
result_merged = pd.concat(result)
del result # clear memory
pd.DataFrame.to_csv(result_merged, "matrix_to_haplotype-chr1n2.txt", sep='t', header=True, index=False)
print()
print('completed all process in "%s" sec. ' % (time.time() - time01))
print('Global maximum memory usage: %.2f (mb)' % current_mem_usage())
print()
'''function to convert the dataframe from genome matrix to desired output '''
def matrix_to_vcf(matrix_df):
print()
time02 = time.time()
# index position of the samples in genome matrix file
sample_idx = [{'10a': 33, '10b': 18}, {'13a': 3, '13b': 19},
{'14a': 20, '14b': 4}, {'16a': 5, '16b': 21},
{'17a': 6, '17b': 22}, {'23a': 7, '23b': 23},
{'24a': 8, '24b': 24}, {'25a': 25, '25b': 9},
{'26a': 10, '26b': 26}, {'34a': 11, '34b': 27},
{'35a': 12, '35b': 28}, {'37a': 13, '37b': 29},
{'38a': 14, '38b': 30}, {'3a': 31, '3b': 15},
{'8a': 32, '8b': 17}]
# sample index stored as ordered dictionary
sample_idx_ord_list = []
for ids in sample_idx:
ids = collections.OrderedDict(sorted(ids.items()))
sample_idx_ord_list.append(ids)
# for haplotype file
header = ['contig', 'pos', 'ref', 'alt']
# adding some suffixes "PI" to available sample names
for item in sample_idx_ord_list:
ks_update = ''
for ks in item.keys():
ks_update += ks
header.append(ks_update+'_PI')
header.append(ks_update+'_PG_al')
#final variable store the haplotype data
# write the header lines first
haplotype_output = 't'.join(header) + 'n'
# to store the value of parsed the line and update the "PI", "PG" value for each sample
updated_line = ''
# read the piped in data back to text like file
matrix_df = pd.DataFrame.to_csv(matrix_df, sep='t', index=False)
matrix_df = matrix_df.rstrip('n').split('n')
for line in matrix_df:
if line.startswith('CHROM'):
continue
line_split = line.split('t')
chr_ = line_split[0]
ref = line_split[2]
alt = list(set(line_split[3:]))
# remove the alleles "N" missing and "ref" from the alt-alleles
alt_up = list(filter(lambda x: x!='N' and x!=ref, alt))
# if no alt alleles are found, just continue
# - i.e : don't write that line in output file
if len(alt_up) == 0:
continue
#print('nMining data for chromosome/contig "%s" ' %(chr_ ))
#so, we have data for CHR, POS, REF, ALT so far
# now, we mine phased genotype for each sample pair (as "PG_al", and also add "PI" tag)
sample_data_for_vcf = []
for ids in sample_idx_ord_list:
sample_data = []
for key, val in ids.items():
sample_value = line_split[val]
sample_data.append(sample_value)
# now, update the phased state for each sample
# also replacing the missing allele i.e "N" and "-" with ref-allele
sample_data = ('|'.join(sample_data)).replace('N', ref).replace('-', ref)
sample_data_for_vcf.append(str(chr_))
sample_data_for_vcf.append(sample_data)
# add data for all the samples in that line, append it with former columns (chrom, pos ..) ..
# and .. write it to final haplotype file
sample_data_for_vcf = 't'.join(sample_data_for_vcf)
updated_line = 't'.join(line_split[0:3]) + 't' + ','.join(alt_up) +
't' + sample_data_for_vcf + 'n'
haplotype_output += updated_line
del matrix_df # clear memory
print('completed haplotype preparation for chromosome/contig "%s" '
'in "%s" sec. ' %(chr_, time.time()-time02))
print('tWorker maximum memory usage: %.2f (mb)' %(current_mem_usage()))
# return the data back to the pool
return pd.read_csv(io.StringIO(haplotype_output), sep='t')
''' to monitor memory '''
def current_mem_usage():
return resource.getrusage(resource.RUSAGE_SELF).ru_maxrss / 1024.
if __name__ == '__main__':
main()
обновление для охотников:
я достиг многопроцессорной обработки с помощью Pool.map() но код вызывает большую нагрузку на память (входной тестовый файл ~ 300 МБ, но нагрузка на память составляет около 6 ГБ). Я был только ожидание нагрузки памяти 3 * 300 МБ при макс.
- может кто-нибудь объяснить, что вызывает такое огромное требование памяти для такого маленького файла и для такого небольшого вычисления длины.
- также, я пытаюсь взять ответ и использовать его, чтобы улучшить это в моей большой программе. Таким образом, добавление любого метода, модуля, который не изменяет структуру вычислительной части (процесс, связанный с процессором), должно быть прекрасным.
- Я включил два тестовых файлов в тестовых целях поиграть с кодом.
- прикрепленный код является полным кодом, поэтому он должен работать так, как он предназначен При копировании-вставке. Любые изменения следует использовать только для улучшения оптимизации на этапах многопроцессорной обработки.
4 ответов
обязательное условие
-
в Python (в следующем я использую 64-битную сборку Python 3.6.5) все является объектом. Это имеет свои накладные расходы и с
getsizeofмы можем видеть точно размер объекта в байтах:>>> import sys >>> sys.getsizeof(42) 28 >>> sys.getsizeof('T') 50 - при использовании системного вызова fork (по умолчанию *nix, см.
multiprocessing.get_start_method()) чтобы создать дочерний процесс, физическая память родителя не копируется и копирование при записи техника. - Fork дочерний процесс по-прежнему будет сообщать полный RSS (размер резидентного набора) родительского процесса. Из-за этого факта, PSS (пропорциональный размер набора) является более подходящей метрикой для оценки использования памяти приложения разветвления. Вот пример со страницы:
- процесс A имеет 50 Кб неразделенной памяти
- процесс B имеет 300 КБ неразделенной памяти
- и процесс A и процесс B имеют 100 КИБ той же области общей памяти
поскольку PSS определяется как сумма неразделенной памяти процесса и доля памяти, совместно используемой с другими процессами, PSS для этих двух процессов являются следующими:
- PSS процесса A = 50 КИБ + (100 КИБ / 2) = 100 КИБ
- PSS процесса B = 300 KiB + (100 KiB / 2) = 350 KiB
фрейм данных
не будем смотреть на ваши
таким образом, мы можем объяснить наивную оценку 7,93 гиб, как:
>>> rows = 4 * 10 ** 6
>>> int_cols = 2
>>> str_cols = 32
>>> int_size = 8
>>> str_size = 58
>>> ptr_size = 8
>>> (int_cols * int_size + str_cols * (str_size + ptr_size)) * rows / 2 ** 30
7.927417755126953
обратите внимание, что str_size составляет 58 байт, а не 50, как мы видели выше для 1-символьный литерал. Это потому что PEP 393 определяет компактные и некомпактные строки. Вы можете проверить это с помощью sys.getsizeof(gen_matrix_df.REF[0]).
фактическое потребление памяти должно быть ~1 гиб, как сообщается gen_matrix_df.info(), это в два раза больше. Мы можем предположить, что это имеет какое-то отношение к выделению памяти (pre), выполненному пандами или NumPy. Следующий эксперимент показывает, что это не без причины (несколько запусков показывают изображение сохранения):
Line # Mem usage Increment Line Contents
================================================
8 53.1 MiB 53.1 MiB @profile
9 def main():
10 53.1 MiB 0.0 MiB with open("genome_matrix_header.txt") as header:
11 53.1 MiB 0.0 MiB header = header.read().rstrip('\n').split('\t')
12
13 2070.9 MiB 2017.8 MiB gen_matrix_df = pd.read_csv('genome_matrix_final-chr1234-1mb.txt', sep='\t', names=header)
14 2071.2 MiB 0.4 MiB gen_matrix_df = gen_matrix_df.drop(columns=[gen_matrix_df.keys()[0]])
15 2071.2 MiB 0.0 MiB gen_matrix_df = gen_matrix_df.drop(columns=[gen_matrix_df.keys()[0]])
16 2040.7 MiB -30.5 MiB gen_matrix_df = gen_matrix_df.drop(columns=[random.choice(gen_matrix_df.keys())])
...
23 1827.1 MiB -30.5 MiB gen_matrix_df = gen_matrix_df.drop(columns=[random.choice(gen_matrix_df.keys())])
24 1094.7 MiB -732.4 MiB gen_matrix_df = gen_matrix_df.drop(columns=[random.choice(gen_matrix_df.keys())])
25 1765.9 MiB 671.3 MiB gen_matrix_df = gen_matrix_df.drop(columns=[random.choice(gen_matrix_df.keys())])
26 1094.7 MiB -671.3 MiB gen_matrix_df = gen_matrix_df.drop(columns=[random.choice(gen_matrix_df.keys())])
27 1704.8 MiB 610.2 MiB gen_matrix_df = gen_matrix_df.drop(columns=[random.choice(gen_matrix_df.keys())])
28 1094.7 MiB -610.2 MiB gen_matrix_df = gen_matrix_df.drop(columns=[random.choice(gen_matrix_df.keys())])
29 1643.9 MiB 549.2 MiB gen_matrix_df = gen_matrix_df.drop(columns=[random.choice(gen_matrix_df.keys())])
30 1094.7 MiB -549.2 MiB gen_matrix_df = gen_matrix_df.drop(columns=[random.choice(gen_matrix_df.keys())])
31 1582.8 MiB 488.1 MiB gen_matrix_df = gen_matrix_df.drop(columns=[random.choice(gen_matrix_df.keys())])
32 1094.7 MiB -488.1 MiB gen_matrix_df = gen_matrix_df.drop(columns=[random.choice(gen_matrix_df.keys())])
33 1521.9 MiB 427.2 MiB gen_matrix_df = gen_matrix_df.drop(columns=[random.choice(gen_matrix_df.keys())])
34 1094.7 MiB -427.2 MiB gen_matrix_df = gen_matrix_df.drop(columns=[random.choice(gen_matrix_df.keys())])
35 1460.8 MiB 366.1 MiB gen_matrix_df = gen_matrix_df.drop(columns=[random.choice(gen_matrix_df.keys())])
36 1094.7 MiB -366.1 MiB gen_matrix_df = gen_matrix_df.drop(columns=[random.choice(gen_matrix_df.keys())])
37 1094.7 MiB 0.0 MiB gen_matrix_df = gen_matrix_df.drop(columns=[random.choice(gen_matrix_df.keys())])
...
47 1094.7 MiB 0.0 MiB gen_matrix_df = gen_matrix_df.drop(columns=[random.choice(gen_matrix_df.keys())])
я хочу закончить этот раздел цитатой из свежие статьи про вопросы дизайна и будущие Pandas2 оригинальным автором панд.
панды эмпирическое правило: иметь от 5 до 10 раз больше ОЗУ, чем размер вашего набора данных
дерево процессов
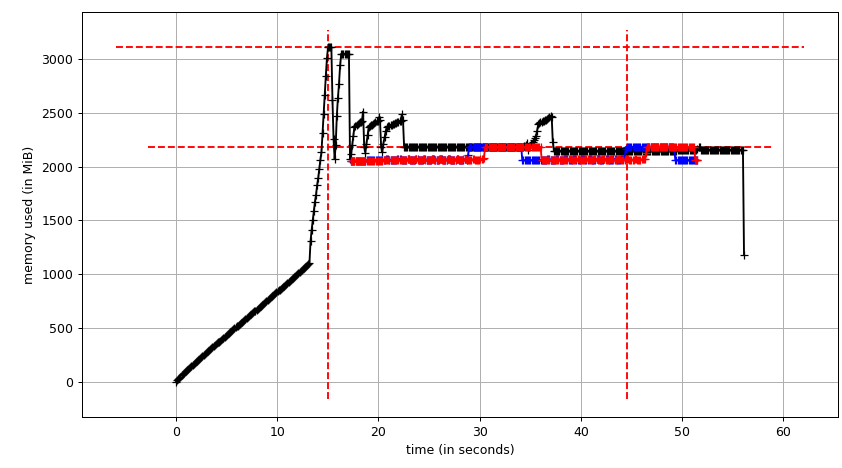
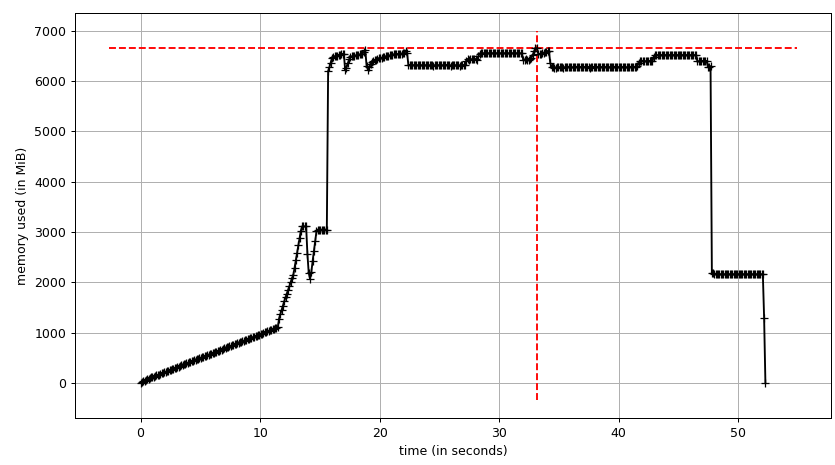
давайте, наконец, подойдем к пулу и посмотрим, можно ли использовать copy-on-write. Мы будем использовать smemstat (доступная форма репозитория Ubuntu) для оценки совместного использования памяти группы процессов и glances записать общесистемный свободная память. Оба могут писать JSON.
мы запустим оригинальный сценарий с Pool(2). Нам понадобится 3 терминальных окна.
-
smemstat -l -m -p "python3.6 script.py" -o smemstat.json 1 glances -t 1 --export-json glances.jsonmprof run -M script.py
затем mprof plot выдает:
график суммы (mprof run --nopython --include-children ./script.py) выглядит так:
обратите внимание, что две диаграммы выше показывают RSS. Этот гипотеза заключается в том, что из-за копирования на запись это не отражает фактическое использование памяти. Теперь у нас есть два файла JSON из smemstat и glances. Я буду следующий скрипт для скрытия файлов JSON в CSV.
#!/usr/bin/env python3
import csv
import sys
import json
def smemstat():
with open('smemstat.json') as f:
smem = json.load(f)
rows = []
fieldnames = set()
for s in smem['smemstat']['periodic-samples']:
row = {}
for ps in s['smem-per-process']:
if 'script.py' in ps['command']:
for k in ('uss', 'pss', 'rss'):
row['{}-{}'.format(ps['pid'], k)] = ps[k] // 2 ** 20
# smemstat produces empty samples, backfill from previous
if rows:
for k, v in rows[-1].items():
row.setdefault(k, v)
rows.append(row)
fieldnames.update(row.keys())
with open('smemstat.csv', 'w') as out:
dw = csv.DictWriter(out, fieldnames=sorted(fieldnames))
dw.writeheader()
list(map(dw.writerow, rows))
def glances():
rows = []
fieldnames = ['available', 'used', 'cached', 'mem_careful', 'percent',
'free', 'mem_critical', 'inactive', 'shared', 'history_size',
'mem_warning', 'total', 'active', 'buffers']
with open('glances.csv', 'w') as out:
dw = csv.DictWriter(out, fieldnames=fieldnames)
dw.writeheader()
with open('glances.json') as f:
for l in f:
d = json.loads(l)
dw.writerow(d['mem'])
if __name__ == '__main__':
globals()[sys.argv[1]]()
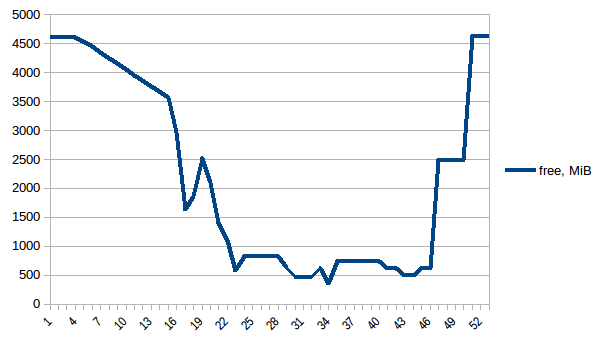
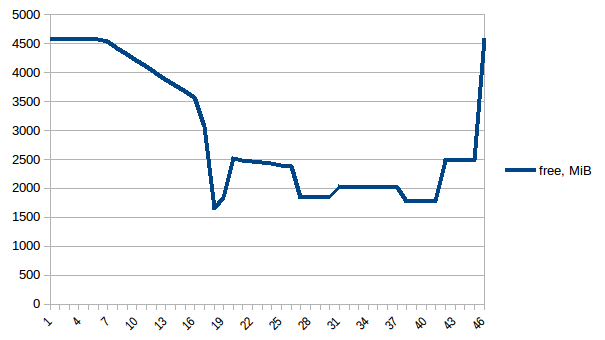
сначала давайте посмотрим на free память.
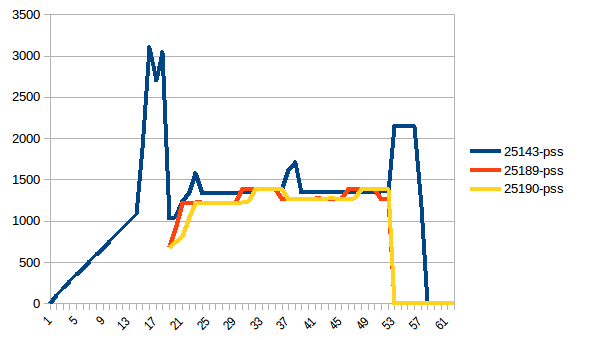
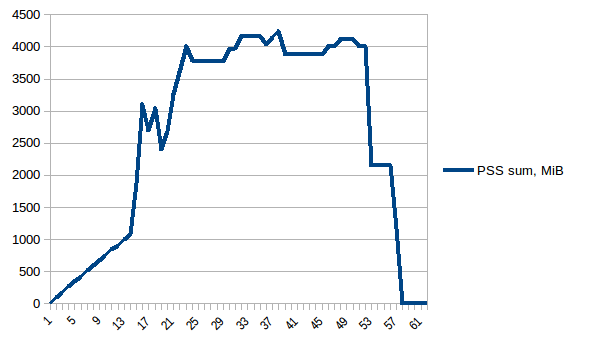
разница между первым и минимум ~4.15 Гб. И вот как выглядят цифры PSS например:
и суммы:
таким образом, мы можем видеть, что из-за копирования на запись фактическое потребление памяти составляет ~4.15 GiB. Но мы все еще сериализуем данные, чтобы отправить их рабочим процессам через Pool.map. Можем ли мы использовать copy-on-write здесь?
Общие сведения
чтобы использовать copy-on-write нам нужно иметь list(gen_matrix_df_list.values()) доступно глобально так работник после вилки все еще может прочитать его.
-
Давайте изменим код после
del gen_matrix_dfнаmainследующим образом:... global global_gen_matrix_df_values global_gen_matrix_df_values = list(gen_matrix_df_list.values()) del gen_matrix_df_list p = Pool(2) result = p.map(matrix_to_vcf, range(len(global_gen_matrix_df_values))) ... - удалить
del gen_matrix_df_listэто будет позже. -
и изменить первые строки
matrix_to_vcfкак:def matrix_to_vcf(i): matrix_df = global_gen_matrix_df_values[i]
теперь давайте повторим его. Свободная память:
не оказывает влияния в данном конкретном случае.обновление
альтернативой обмену данными без копирования при записи может быть делегирование его ядру с самого начала с помощью numpy.memmap. Вот!--275-->пример реализации С высокая производительность обработки данных в Python поговорить. The сложно затем, чтобы сделать панд использовать mmaped массив Numpy.
у меня была та же проблема. Мне нужно было обработать огромный текстовый корпус, сохраняя при этом базу знаний из нескольких кадров данных миллионов строк, загруженных в память. Я думаю, что эта проблема распространена, поэтому я буду держать свой ответ ориентированным на общие цели.
A сочетание из настроек решена проблема для меня (1 & 3 & 5 только может сделать это для вас):
использовать
Pool.imap(илиimap_unordered) вместоPool.map. Это будет перебирать данные лениво, чем загрузка всего этого в память перед началом обработки.установите значение
при использовании multiprocessing.Pool ряд дочерних процессов будет создан с помощью fork() системный вызов. Каждый из этих процессов начинается с точной копии памяти родительского процесса в то время. Потому что вы загружаете csv перед созданием Pool размера 3, Каждый из этих 3 процессов в пуле будет излишне иметь копию фрейма данных. (gen_matrix_df а также gen_matrix_df_list будет существовать в нынешнем процессе, а также в каждом из 3 дочерних процессов, поэтому 4 копии каждая из этих структур будет в памяти)
попробуйте создать Pool перед загрузкой файла (в самом начале), который должен уменьшить использование памяти.
если он все еще слишком высок, вы можете:
-
дамп gen_matrix_df_list в файл, 1 элемент в строке, e.g:
import os import cPickle with open('tempfile.txt', 'w') as f: for item in gen_matrix_df_list.items(): cPickle.dump(item, f) f.write(os.linesep) -
использовать
Pool.imap()на итераторе по строкам, которые вы сбросили в этот файл, например:with open('tempfile.txt', 'r') as f: p.imap(matrix_to_vcf, (cPickle.loads(line) for line in f))(обратите внимание, что
matrix_to_vcfпринимает(key, value)Кортеж в приведенном выше примере, а не просто значение)
надеюсь, это поможет.
NB: я не тестировал код выше. Это только для демонстрации идеи.
ОБЩИЙ ОТВЕТ О ПАМЯТИ С МНОГОПРОЦЕССОРНОЙ
вы спросили:"Что заставляет выделяться столько памяти". Ответ состоит из двух частей.
первый, как вы уже заметили, каждого multiprocessing работник получает собственную копию данных (ЦИТ.отсюда), поэтому вы должны разбить большие аргументы. Или для больших файлов, прочитайте их немного за раз, если это возможно.
By по умолчанию работники пула-это реальные процессы Python, разветвленные использование модуля многопроцессорной обработки стандартной библиотеки Python при n_jobs != 1. Аргументы, передаваемые в качестве входных данных для параллельного вызова сериализовано и перераспределено в памяти каждого рабочего процесса.
Это может быть проблематично для больших аргументов, как они будут перераспределены n_jobs раз рабочими.
второй, если вы пытаетесь освободить память, вам нужно чтобы понять, что Python работает иначе, чем другие языки, и вы полагаетесь на del, чтобы освободить память, когда это не. Я не знаю, лучше ли это, но в моем собственном коде я преодолел это, переназначив переменную в None или пустой объект.
ДЛЯ ВАШЕГО КОНКРЕТНОГО ПРИМЕРА-МИНИМАЛЬНОЕ РЕДАКТИРОВАНИЕ КОДА
пока вы можете поместить свои большие данные в память два раза, Я думаю, вы можете сделать то, что вы пытаетесь сделать это, просто изменив одну строку. Я написал очень похожий код, и он работал для меня, когда я переназначил переменную (вице-вызов del или любой сбор мусора). Если это не сработает, вам может потребоваться следовать приведенным выше рекомендациям и использовать дисковый ввод-вывод:
#### earlier code all the same
# clear memory by reassignment (not del or gc)
gen_matrix_df = {}
'''Now, pipe each dataframe from the list using map.Pool() '''
p = Pool(3) # number of pool to run at once; default at 1
result = p.map(matrix_to_vcf, list(gen_matrix_df_list.values()))
#del gen_matrix_df_list # I suspect you don't even need this, memory will free when the pool is closed
p.close()
p.join()
#### later code all the same
ДЛЯ ВАШЕГО КОНКРЕТНОГО ПРИМЕРА-ОПТИМАЛЬНОЕ ИСПОЛЬЗОВАНИЕ ПАМЯТИ
пока вы можете поместить свои большие данные в память после, и Вы имеете некоторое представление о том, насколько велик ваш файл, вы можете использовать панды read_csv частичное чтение файла, читать только nrows за раз если вы действительно хотите, чтобы контролировать, сколько данных можно прочитать В, или [фиксированный объем памяти с помощью chunksize], который возвращает итератор5. Под этим я подразумеваю, что параметр nrows-это всего лишь одно чтение: вы можете использовать его, чтобы просто заглянуть в файл, или если по какой-то причине вы хотите, чтобы каждая часть имела точно такое же количество строк (потому что, для например, если какие-либо из ваших данных являются строками переменной длины, каждая строка не будет занимать одинаковый объем памяти). Но я думаю, что для целей подготовки файла к многопроцессорной обработке будет намного проще использовать куски, потому что это напрямую связано с памятью, что вас беспокоит. Будет проще использовать trial & error, чтобы вписаться в память на основе определенных кусков размера, чем количество строк, что изменит объем использования памяти в зависимости от того, сколько данных находится в строках. Единственный трудная часть заключается в том, что по какой-то конкретной причине приложения вы группируете некоторые строки, поэтому это просто делает его немного сложнее. Использование кода в качестве примера:
'''load the genome matrix file onto pandas as dataframe.
This makes is more easy for multiprocessing'''
# store the splitted dataframes as list of key, values(pandas dataframe) pairs
# this list of dataframe will be used while multiprocessing
#not sure why you need the ordered dict here, might add memory overhead
#gen_matrix_df_list = collections.OrderedDict()
#a defaultdict won't throw an exception when we try to append to it the first time. if you don't want a default dict for some reason, you have to initialize each entry you care about.
gen_matrix_df_list = collections.defaultdict(list)
chunksize = 10 ** 6
for chunk in pd.read_csv(genome_matrix_file, sep='\t', names=header, chunksize=chunksize)
# now, group the dataframe by chromosome/contig - so it can be multiprocessed
gen_matrix_df = chunk.groupby('CHROM')
for chr_, data in gen_matrix_df:
gen_matrix_df_list[chr_].append(data)
'''Having sorted chunks on read to a list of df, now create single data frames for each chr_'''
#The dict contains a list of small df objects, so now concatenate them
#by reassigning to the same dict, the memory footprint is not increasing
for chr_ in gen_matrix_df_list.keys():
gen_matrix_df_list[chr_]=pd.concat(gen_matrix_df_list[chr_])
'''Now, pipe each dataframe from the list using map.Pool() '''
p = Pool(3) # number of pool to run at once; default at 1
result = p.map(matrix_to_vcf, list(gen_matrix_df_list.values()))
p.close()
p.join()